- az optima becslése az Ornstein-Uhlenbeck evolúció alatt
- Michael R. May
- Utolsó módosítás: szeptember 16, 2019
- az evolúciós Optima becslése
- Ornstein-Uhlenbeck modell
- olvassa el az adatokat
- a modell megadása
- fa modell
- Rate paraméter
- adaptációs paraméter
- Optimum
- Ornstein-Uhlenbeck modell
- MCMC analízis futtatása
- monitorok meghatározása
- az MCMC szimuláció inicializálása és futtatása
- gyakorlat 1
- az OU és a BM modellek összehasonlítása
- Modellválasztás reverzibilis ugrással MCMC
- reverzibilis ugrás OU és BM modellek között
az optima becslése az Ornstein-Uhlenbeck evolúció alatt
Michael R. May
Utolsó módosítás: szeptember 16, 2019
az evolúciós Optima becslése
ez az oktatóanyag bemutatja, hogyan lehet meghatározni egy Ornstein-Uhlenbeck modellt, ahol feltételezzük, hogy az optimális fenotípus állandó az időben kalibrált filogenitás (hiányzó referencia) ágai között a gerinces kládok (hiányzó referencia) (log) testméretének adatkészletei felhasználásával. Az oktatóanyag minden egyes összetevőjének valószínűségi grafikus modell-ábrázolását biztosítjuk. A modell megadása után megbecsülheti az Ornstein-Uhlenbeck evolúció paramétereit a Markov chain Monte Carlo (MCMC) segítségével.
Ornstein-Uhlenbeck modell
az egyszerű Ornstein-Uhlenbeck (OU) modell alatt feltételezzük, hogy a folytonos karakter az optimális érték, a $\theta$felé fejlődik. A karakter sztochasztikusan fejlődik egy drift paraméter szerint, $ \ sigma^2$. A karaktert az optimális felé húzza az alkalmazkodási sebesség, $ \ alpha$; az alfa nagyobb értékei azt jelzik, hogy a karakter erősebben húzódik a $\theta$felé. Amint a karakter elmozdul a $\theta$ – tól, a $\alpha$ paraméter határozza meg, hogy a karakter milyen erősen visszahúzódik. Emiatt a $ \ alpha$ – t néha “gumiszalag” paraméternek nevezik. Amikor a $\alpha = 0$ adaptációs paraméter sebessége, az OU modell összeomlik a BM modellre. Az így kapott grafikus modell meglehetősen egyszerű, mivel a folytonos karakterek valószínűsége csak a filogenitástól függ (amiről feltételezzük, hogy ismert ebben az oktatóanyagban) és a három OU paramétertől ().
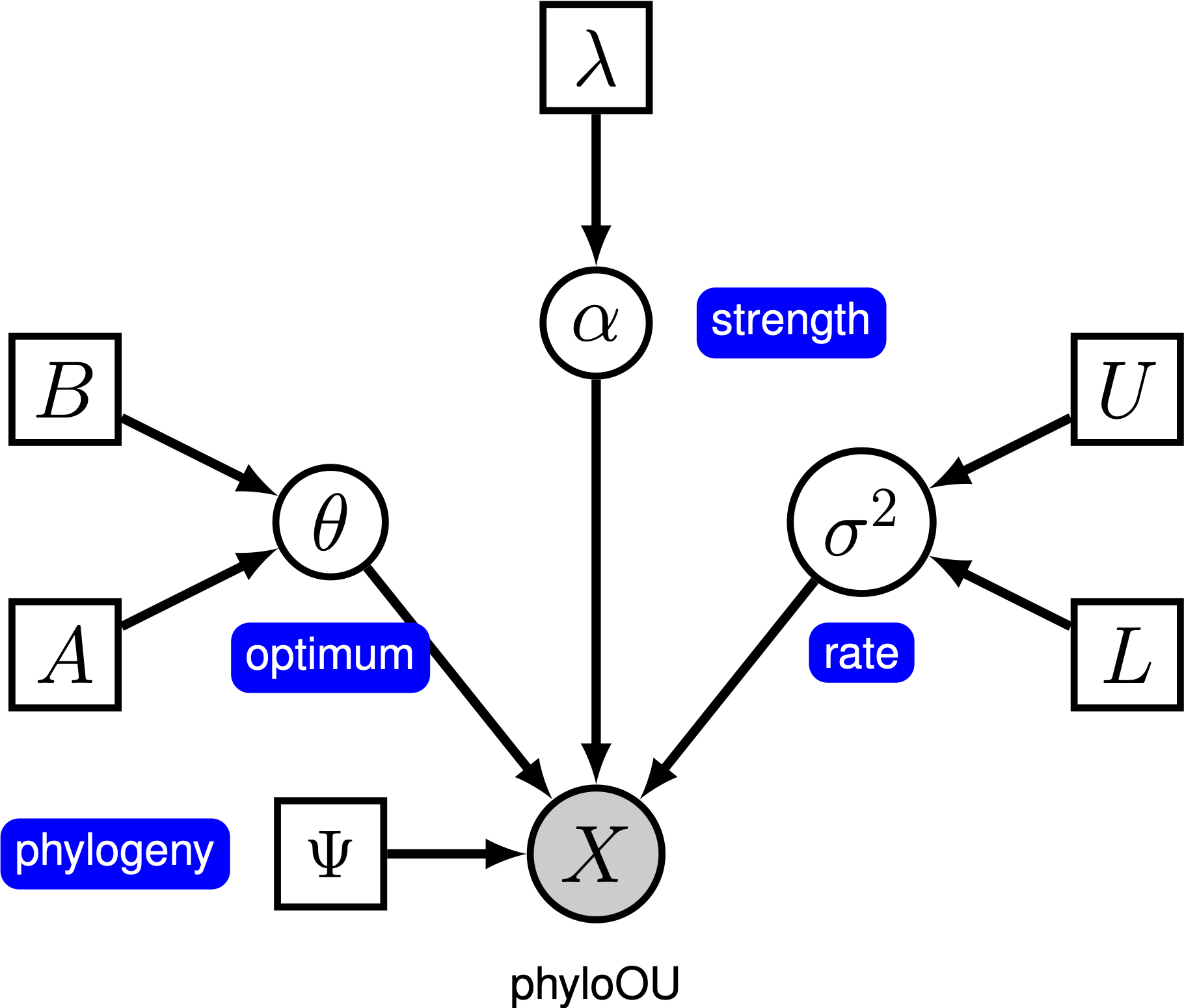
ebben az oktatóanyagban a 66 gerinces törzsfejlődést és (log) testméret-adatkészletet használjuk (Landis and Schraiber 2017).
a teljes OU-modell specifikáció a mcmc_OU.Rev nevű fájlban található.
olvassa el az adatokat
először eldöntjük, hogy a 66 gerinces adatkészlet közül melyiket használjuk. Itt feltételezzük, hogy az első adatkészletet (Acanthuridae) elemezzük, de nyugodtan válasszon bármelyik adatkészletet.
dataset <- 1most a választott adatkészletünknek megfelelő (időben kalibrált) fában olvasunk.
T <- readTrees("data/trees.nex")ezután ugyanazon adatkészlet karakteradatait olvassuk el.
data <- readContinuousCharacterData("data/traits.nex")ezenkívül inicializálunk egy változót a vektorunkhoz ofmoves és monitorok:
moves = VectorMoves()monitors = VectorMonitors()a modell megadása
fa modell
ebben az oktatóanyagban feltételezzük, hogy a fa terület nélkül ismert. Létrehozunk egy állandó csomópontot a fa számára, amely megfelel a megfigyelt filogenitásnak.
tree <- TRate paraméter
az evolúció sztochasztikus sebességét a rate paraméter, $\sigma^2$szabályozza. A rate paramétert egy logból rajzoljukegységes prior. Ez a prior egységes a log skálán, ami azt jelenti, hogy az jelentése tudatlanság az arány nagyságrendjével kapcsolatban.
sigma2 ~ dnLoguniform(1e-3, 1)annak érdekében, hogy megbecsüljük a $\sigma^2$ hátsó eloszlását, meg kell adnunk egy MCMC javaslat mechanizmust, amely ezen a csomóponton működik. Mivel a $ \ sigma^2$ egy rate paraméter, és ezért pozitívnak kell lennie, egy mvScalenevű skálázási lépést használunk.
moves.append( mvScale(sigma2, weight=1.0) )adaptációs paraméter
az optimális adaptáció sebességét a $\alpha$paraméter határozza meg. A $\alpha$ – t egy exponenciális előzetes eloszlásból húzzuk ki, és egy skálajavaslatot helyezünk rá.
alpha ~ dnExponential(10)moves.append( mvScale(alpha, weight=1.0) )Optimum
az optimális értéket egy homályos egységes prior-tól -10-től 10-ig terjedő értékből vonjuk le (ezt a priort meg kell változtatnod, ha a karaktered kívül esik ezen a tartományon). Mivel ez a paraméter lehet pozitív vagy negatív, egy dia mozdulattal javasolunk változtatásokat az MCMC során.
theta ~ dnUniform(-10, 10)moves.append( mvSlide(theta, weight=1.0) )Ornstein-Uhlenbeck modell
most, hogy megadtuk a modell paramétereit, levonhatjuk a karakteradatokat a megfelelő filogenetikai OU modellből. Ebben a példában a reml algoritmust használjuk a valószínűség hatékony kiszámításához (Felsenstein 1985). Feltételezzük, hogy a karakter az optimális értéknél kezdődik a fa gyökerénél.
X ~ dnPhyloOrnsteinUhlenbeckREML(tree, alpha, theta, sigma2^0.5, rootStates=theta)megjegyezve, hogy $X$ a megfigyelt adat (), a data – ot ehhez a sztochasztikus csomóponthoz rögzítjük.
X.clamp(data)végül létrehozunk egy munkaterület-objektumot a teljes modellhez model() – vel. Ne feledje, hogy a munkaterület objektumok inicializálása a = operátorral történik, és maguk nem részei a Bayes-féle grafikus modellnek. A model() függvény bejárja a teljes modellgráfot, és megkeresi a megadott modell összes csomópontját. Ez az objektum kényelmes módot nyújt a teljes modellobjektumra való hivatkozásra, nem csak egyetlen DAG csomópontra.
mymodel = model(theta)MCMC analízis futtatása
monitorok meghatározása
MCMC elemzésünkhöz be kell állítanunk egy monitorvektort a Markov-lánc állapotainak rögzítésére. A monitor funkciók mindegyike mn*, ahol * a helyettesítő karakter, amely a monitor típusát képviseli. Először inicializáljuk a modellmonitort a mnModel funkcióval. Ez létrehoz egy új monitor változót, amely kiadja az összes modellparaméter állapotát, amikor átkerül egy MCMC függvénybe.
monitors.append( mnModel(filename="output/simple_OU.log", printgen=10) )ezenkívül hozzon létre egy képernyőmonitort, amely a megadott változók állapotát mnScreen:
monitors.append( mnScreen(printgen=1000, sigma2, alpha, theta) )az MCMC szimuláció inicializálása és futtatása
egy teljesen meghatározott modellel, egy monitorkészlettel és egy sor mozdulattal, a wecan Most beállíthatja az MCMC algoritmust, amely a paraméterértékeket a hátsó valószínűségük arányában fogja mintavételezni. A mcmc() függvény létrehozza az MCMC objektumunkat:
mymcmc = mcmc(mymodel, monitors, moves, nruns=2, combine="mixed")most futtassa az MCMC-t:
mymcmc.run(generations=50000)amikor az elemzés befejeződött, a megfigyelt fájlok a sajátban lesznek.kimeneti könyvtár.
a Rev fájl ezen elemzés elvégzéséhez: mcmc_OU.Rev
az OU folyamat során kialakuló karakterek egy stacionárius Eloszlás felé hajlanak, amely normális eloszlás átlagos $\theta$ és variancia $\sigma^2 \div 2\alpha$. Ezért, ha az evolúció sebessége magas (vagy a fa ágai viszonylag hosszúak), nehéz lehet külön megbecsülni a $\sigma^2$ és a $\alpha$ értéket, mivel mindkettő meghatározza a folyamat hosszú távú varianciáját. Láthatjuk, hogy ez befolyásolja-e elemzésünket a paraméterek közös hátsó eloszlásának vizsgálatával Tracer. Ha a paraméterek pozitívan korrelálnak, haboznunk kell értelmezni marginális eloszlásaikat (vagyis ne tegyünk külön következtetéseket az adaptáció sebességéről vagy a variancia paraméterről).
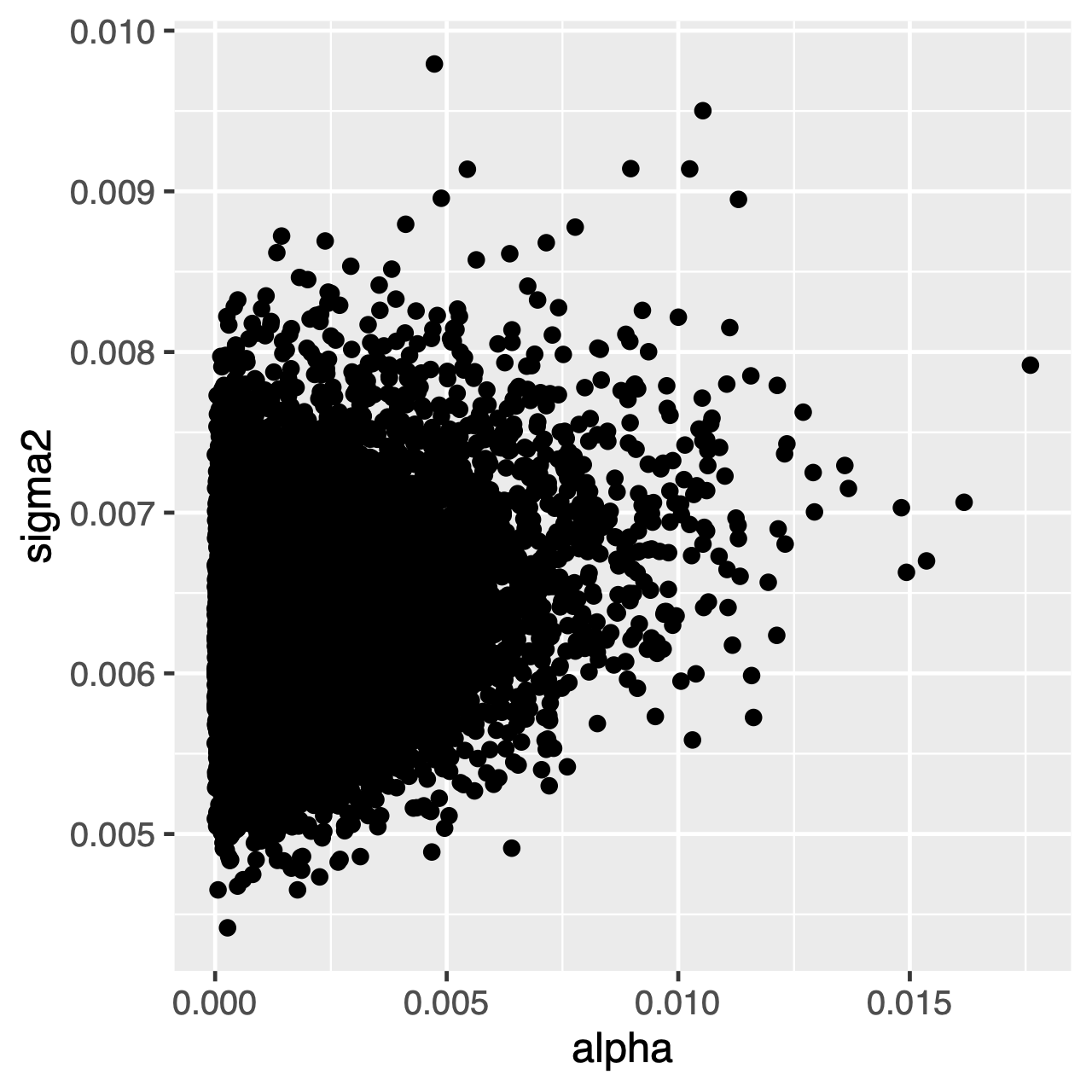
gyakorlat 1
- futtasson egy MCMC szimulációt az OU optimális hátsó eloszlásának becsléséhez (
theta). - töltse be a generált kimeneti fájlt
Tracer: mi az átlagos hátsó becslésthetaés mi a becsült HPD? - a
Tracerhasználatával hasonlítsa össze aalphaés asigma2közös hátsó eloszlását. Ezek a paraméterek korreláltak vagy korrelálatlanok? - hasonlítsa össze az előző átlagot a hátsó átlaggal. (Tipp: Használja a
underPrior=TRUEopcionális argumentumot amymcmc.run()függvényben) különböznek egymástól (pl. )? A hátsó átlag az előző 95% – os valószínűségi intervallumon kívül van?
az OU és a BM modellek összehasonlítása
most, hogy mind a BM, mind az OU modellekhez illeszkedhetünk, természetesen érdemes tudni, hogy melyik modell jobban illeszkedik. Ebben a részben megtanuljuk, hogyan kell használni a reverzibilis Ugrás Markov lánc Monte Carlo összehasonlítani a fit OU és BM modellek.
Modellválasztás reverzibilis ugrással MCMC
annak a hipotézisnek a teszteléséhez, hogy egy karakter szelektív optimum felé fejlődik, két modellt képzelünk el. Az első modell, ahol nincs adaptáció az optimum felé, az az eset, amikor $ \ alpha = 0$. A második modell megfelel az OU modellnek $\alpha > 0$értékkel. Ez azért működik, mert a Brown-mozgás az OU modell speciális esete, amikor az alkalmazkodás sebessége 0. Sajnos, mivel a $ \ alpha$ egy folyamatos paraméter, egy szabványos Markov-lánc soha nem fog meglátogatni olyan államokat,ahol minden érték pontosan 0. Szerencsére használhatjuk a reverzibilis ugrást, hogy a Markov-lánc fontolóra vegye a Brown-mozgás modell meglátogatását. Ez magában foglalja a két modell előzetes valószínűségének megadását, valamint a $\alpha$ előzetes eloszlásának megadását az OU modellhez.
az rjMCMC használata lehetővé teszi a Markov-lánc számára, hogy meglátogassa a két modellt a hátsó valószínűségük arányában. A $i$ modell hátsó valószínűsége egyszerűen a minták töredéke, ahol a lánc meglátogatta ezt a modellt. Mivel a modelleken priort is megadunk, az OU modell Bayes-tényezőjét a következőképpen számíthatjuk ki:
\
ahol $P( \text{OU model} \mid X)$ és $P( \text{OU model})$ az OU modell posterior és prior valószínűsége.
reverzibilis ugrás OU és BM modellek között
az rjMCMC engedélyezéséhez egyszerűen egy reverzibilis ugrást kell elhelyeznünk a megfelelő paraméter, $\alpha$előtt. Módosíthatjuk a prior-t alpha úgy, hogy vagy 0 állandó értéket vegyen fel, vagy egy korábbi eloszlásból származik. Végül megadunk egy előzetes valószínűséget az OU modellen p = 0.5.
alpha ~ dnReversibleJumpMixture(0.0, dnExponential(10), 0.5)ezután egy reverzibilis ugrási javaslatot nyújtunk be alpha, amely változtatásokat javasol a két modell között.
moves.append( mvRJSwitch(alpha, weight=1.0) )ezenkívül megadjuk a normál mvScale javaslatot arra az esetre, amikor az MCMC meglátogatja az OU modellt.
moves.append( mvScale(alpha, weight=1.0) )tartalmaz egy változót, amelynek értéke 1, amikor a lánc az OU modellt látogatja, és egy megfelelő változót, amelynek értéke 1, amikor a BM modellt látogatja. Ez lehetővé teszi számunkra, hogy könnyen kiszámítsuk a modellek hátsó valószínűségét, mert egyszerűen ki kell számolnunk ennek a paraméternek a hátsó középértékét.
is_OU := ifelse(alpha != 0.0, 1, 0)is_BM := ifelse(alpha == 0.0, 1, 0)a minták azon hányada, amelyeknél is_OU = 1 az OU modell hátsó valószínűsége. Alternatív megoldásként ennek a mutatóváltozónak a hátsó átlagos becslése megfelel az OU modell hátsó valószínűségének. Ezek az értékek felhasználhatók a fenti Bayes-faktor egyenletben bármelyik modell Bayes-faktor támogatásának kiszámításához.
- Felsenstein J. 1985. Filogenetika és az összehasonlító módszer. Az Amerikai Természettudós.:1-15.10.1086/284325
- D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O., D. O. 2014. Valószínűségi grafikus Modellábrázolás a Filogenetikában. Szisztematikus Biológia. 63: 753-771. 10.1093/sysbio / syu039
- Landis M. J., Schraiber J. G. 2017. Pulzáló evolúció alakú modern gerinces testméretek. A Nemzeti Tudományos Akadémia Közleményei. 114: 13224-13229.10. 1073 / pnas.1710920114