- Introduction
- La conception optimisée d’ARNsi
- ÉTAPE 1 : Sélection de la séquence d’ARNSI fonctionnelle
- ÉTAPE 2: Sélection d’une séquence d’ARNsi avec des effets hors cible réduits
- ÉTAPE 3: L’élimination de Gènes Hors cible Appariés Presque parfaits
- Logiciel de conception siRNA
- Conclusion
- Déclaration de conflit d’intérêts
- Remerciements
Introduction
L’interférence d’ARN (ARNi) est une technique largement utilisée par laquelle un petit ARN interférent (ARNi) régule à la baisse un gène cible spécifique avec une séquence complémentaire parfaite, et promise à être utilisée dans des applications thérapeutiques pour des maladies humaines (Castanotto et Rossi, 2009; Ketting, 2011). Bien que l’homme possède plus de 20 000 gènes, il est souhaitable d’utiliser un ARNsi qui est hautement fonctionnel et n’a aucun effet sur d’autres gènes que sa cible spécifique. Dans cet article, nous passons en revue une méthode optimisée pour concevoir des ARNsi basés sur le mécanisme de l’ARNi. De plus, nous présentons les sites Web ouverts au public pour la sélection des séquences d’ARNSI.
La conception optimisée d’ARNsi
Duplexes d’ARN 21 nucléotides (nt) avec 2 surplombs nt 3′ (ARNsi) est généralement utilisée pour les expériences d’ARNi. Lors de l’administration dans les cellules, les ARNSI sont incorporés dans le complexe de silençage induit par l’ARN (RISC) sous forme d’ARN double brin. Le RISC est le complexe effecteur contenant la protéine Argonaute (Ago) ayant une activité tranchante (Hammond et al., 2001; Martinez et coll., 2002). Le brin guide siRNA contenant l’extrémité 5′ thermodynamiquement moins stable est préférentiellement retenu par RISC (Khvorova et al., 2003; Schwarz et coll., 2003; Ui-Tei et coll., 2004). Les brins passagers de la plupart des SIRNA double brin chargés sur RISC sont clivés par la protéine Ago2 et dégradés (Matranga et al., 2005; Rand et coll., 2005; Leuschner et coll., 2006). Le brin guide retenu couple l’ARNm cible avec une séquence parfaitement complémentaire, et le réprime par clivage par la protéine Ago2 en position nucléotidique 10 du brin guide siRNA (Elbashir et al., 2001; Hammond et coll., 2001; Martinez et coll., 2002). Cependant, une preuve accumulée à partir d’expériences à l’échelle du génome indique qu’un grand nombre d’ARNM présentant une complémentarité partielle avec le brin guide sont également réduits (Jackson et al., 2003, 2006; Lim et coll., 2005; Birmingham et coll., 2006; Ui-Tei et coll., 2008). Ce phénomène est appelé effet hors cible dépendant de la graine et de préférence observé dans les ARNm 3′ UTRs. Le mécanisme de reconnaissance cible de cet effet hors cible est connu pour être similaire à celui du silençage génique médié par les miARN (Lewis et al., 2005; Lim et coll., 2005; Grimson et coll., 2007). Les transcrits avec des séquences complémentaires à la région de germination positionnée 2-8 à partir du terminal 5′ sont principalement réduits. La région de germination est connue pour être située à la surface d’Ago sous une forme quasi hélicoïdale pour servir de site d’entrée ou de nucléation pour de petits ARN dans les RISC (Ma et al., 2005; Yuan et coll., 2005). Ainsi, la région de graine identifie d’abord les ARNm cibles, puis forme un appariement de base parfait avec l’ARNm cible cible et induit l’ARNi par Ago2.
Sur la base du mécanisme de l’ARNi, un ARNsi spécifique au gène cible est considéré comme sélectionnable selon les trois étapes suivantes.
ÉTAPE 1 : Sélection de la séquence d’ARNSI fonctionnelle
L’efficacité de knockdown des ARNSI dépend fortement de leurs séquences. Nous avons préconisé la règle empirique qui prescrit les caractéristiques des ARNSI hautement fonctionnels (Ui-Tei et al., 2004), une telle règle est appelée règle Ui-Tei (figure 1). Le siRNA sélectionné par la règle Ui-Tei satisfait simultanément aux quatre conditions suivantes: (1) A ou U à la position 1 à partir de l’extrémité 5 ‘ du brin guide siRNA, (2) G ou C à la position 19, (3) richesse AU (AU ≥4) dans les positions 1 à 7, et (4) pas de long étirement GC ≥10. À l’exception de (4), notre règle indiquait que le siRNA fonctionnel a une stabilité asymétrique dans les terminaux 5′ et 3’. Notre validation expérimentale à l’aide du test de rapport de la luciférase a montré que 98% des siRNA satisfaisant aux conditions ci-dessus réduisaient l’expression du rapporteur de la luciférase en dessous de 33% (Ui-Tei et al., 2004). D’autres groupes ont également démontré les règles des SIRNA hautement fonctionnels appelées règle de Reynolds (Reynolds et al., 2004) et la règle d’Amarzguioui (Amarzguioui et Prydz, 2004) comme résumé à la figure 1. Ces règles ont également clairement montré que les SIRNA fonctionnels sont asymétriques : un brin d’ARN à terminal 5′ instable était efficace comme brin guide. De plus, commun dans ces règles, l’extrémité 5′ du brin de guidage d’ARNsi fonctionnel était préférable à A ou U. Il a été révélé plus tard que les résultats reflétaient les caractéristiques structurelles de l’Ago2 humain (Frank et al., 2010). La structure cristalline d’un domaine MOYEN (moyen) provenant d’expériences de titrage AGO2 et RMN humaines a montré que les monophosphates nucléotidiques, AMP et UMP, se lient avec une affinité jusqu’à 30 fois plus élevée que la CMP ou la GMP, fournissant des preuves structurelles des interactions spécifiques aux nucléotides dans le domaine MOYEN des protéines eucaryotes AGO.
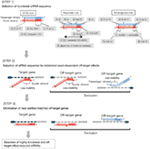
Figure 1. Représentation schématique pour la sélection des SIRNA fonctionnels et hors cible à effet réduit. Sélection des siRNA hautement fonctionnels par les règles Ui-Tei, Reynolds, Amarzguioui, ou leur combinaison (ÉTAPE 1). Sélection d’ARNSI à faible stabilité dans les duplex cibles de semences (ÉTAPE 2). Élimination des ARNSI avec des séquences appariées presque parfaites aux gènes non cibles (ÉTAPE 3). Dans chaque règle, la position des nucléotides indique le nombre de nucléotides comptés à partir de la borne 5′ du brin guide.  :
:  A/U à la position 1.
A/U à la position 1.  G/C à la position 19.
G/C à la position 19.  4 à 7 A/ Us dans les positions 1 à 7.
4 à 7 A/ Us dans les positions 1 à 7. 
 :
:  Teneur en GC (30% -52%).
Teneur en GC (30% -52%).  A/U ≥ 3 aux positions 1-5.
A/U ≥ 3 aux positions 1-5.  Absence de répétitions internes.
Absence de répétitions internes.  A à la position 1.
A à la position 1.  A à la position 17.
A à la position 17.  U à la position 10.
U à la position 10.  Pas G/C à la position 1.
Pas G/C à la position 1. 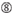 Pas G à la position 7.
Pas G à la position 7. 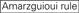 :
:  Une asymétrie dans la stabilité des extrémités duplex (mesurée comme le différentiel A/U des trois paires de base terminales à chaque extrémité du duplex).
Une asymétrie dans la stabilité des extrémités duplex (mesurée comme le différentiel A/U des trois paires de base terminales à chaque extrémité du duplex).  G ou C à la position 19.
G ou C à la position 19.  A ou U à la position 1.
A ou U à la position 1.  A ou U à la position 14.
A ou U à la position 14.  Pas U à la position 19.
Pas U à la position 19.  Pas G à la position 1.
Pas G à la position 1.
ÉTAPE 2: Sélection d’une séquence d’ARNsi avec des effets hors cible réduits
Pour éviter les effets hors cible dépendants de la graine, une approche peut consister à sélectionner le brin guide d’ARNsi dont la séquence de graine n’est complémentaire d’aucune séquence dans le 3′ UTR de tous les gènes non ciblés. Cependant, cette approche s’est avérée impossible, car les ARNM humains avec la séquence de semences à sept NT la plus rare présentaient encore des complémentarités de semences avec plusieurs ARNM non ciblés. Ainsi, nous avons cherché les règles qui régissent la capacité des ARNSI à induire un effet hors cible dépendant de la graine, et avons révélé que l’efficacité de l’effet hors cible est fortement corrélée à la stabilité thermodynamique du duplex formé entre la région de graine du brin guide d’ARNsi et son ARNm cible (Ui-Tei et al., 2008). La température de fusion (Tm), l’un des paramètres thermodynamiques pour la formation du duplex d’ARN, a montré une forte corrélation positive avec l’induction d’effets hors cible dépendants de la graine. Ainsi, la sélection des siRNA avec une faible Tm du duplex graine-cible devrait minimiser le silence hors cible dépendant de la graine (figure 1). La Tm de 21,5 ° C peut servir de référence, ce qui distingue les séquences de graines presque hors cible des séquences positives hors cible. De plus, l’effet hors cible pouvant être provoqué non seulement par le brin de guidage mais aussi par le brin passager, les SIRNA dont la cible de germination Tm est suffisamment faible pour les deux brins sont favorables.
ÉTAPE 3: L’élimination de Gènes Hors cible Appariés Presque parfaits
Même lorsque la valeur Tm du duplex graine-cible est suffisamment faible, le silençage des gènes cibles peut toujours avoir lieu si la région non graine est complètement complémentaire. Par conséquent, à la troisième étape, les ARNR qui correspondent presque parfaitement à tout autre relevé de notes non ciblé ont été éliminés (figure 1).
Logiciel de conception siRNA
Nous avons présenté le logiciel de conception siRNA, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figure 2), qui fournit un logiciel de conception d’ARNsi fonctionnel et spécifique à la cible selon les procédures mentionnées ci-dessus (Naito et al., 2009). Dans le paramètre par défaut, siRNAs satisfaisant la règle Ui-Tei peut être sélectionnée. Lorsque les siRNA fonctionnels candidats peuvent former des duplexes cibles avec des valeurs de Tm inférieures à 21,5 ° C, et que leurs régions 19-nt couvrant les positions 2 à 20 des deux brins présentent au moins deux non-appariements avec tout autre transcrit non ciblé, siDirect 2.0 peut concevoir au moins un siRNA qualifié pour > 94% des séquences d’ARNm humaines dans RefSeq.

Figure 2. Vues d’écran du logiciel de conception siRNA siDirect 2.0. A) Page du haut (http://siDirect2.RNAi.jp/). B) Paramètres facultatifs pour la conception de l’ARNSI. (C) Page de résultats. D) Liste détaillée des candidats hors cible avec des correspondances presque parfaites. L’alignement entre chaque transcription hors cible et la séquence d’ARNSI permet de visualiser les positions des non-concordances.
D’autres logiciels permettant de sélectionner des SIRNA fonctionnels étaient ouverts au public, comme le montre le tableau 1. Dans beaucoup d’entre eux, la règle Ui-Tei (Ui-Tei et al., 2004), règle de Reynolds (Reynolds et al., 2004), la règle d’Amarzguioui (Amarzguioui et Prydz, 2004), la règle de Tuschl (Elbashir et al., 2002), et leur combinaison étaient fréquemment et largement utilisées. Pour éliminer les gènes non cibles appariés presque parfaits, BLAST search a été utilisé pour la recherche d’homologie dans plusieurs logiciels. Cependant, étant donné que la recherche de SOUFFLE n’est pas aussi précise pour les séquences courtes comme siRNAs, siDirect, WU-BLAST et Bowtie, qui sont des moteurs de recherche d’homologie très précis pour les séquences courtes sont souvent utilisés. Parmi eux, siDirect 2.0 peut apporter les résultats les plus précis. En outre, certains logiciels prennent en compte les fonctionnalités supplémentaires, telles que la structure secondaire de l’ARNm (Ladunga, 2007; Lu et Mathews, 2008), l’épissage alternatif (Park et al., 2008), ou la séquence de motifs par laquelle la réponse immunitaire par un virus à ARN peut se produire (Gong et al., 2008). Ces fonctionnalités ne sont pas prises en compte dans siDirect 2.0. Ainsi, pour prendre en compte ces fonctionnalités, les siRNA couramment sélectionnés par siDirect 2.0 et les autres logiciels appropriés peuvent produire un résultat optimal.
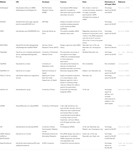
Tableau 1. Logiciels de conception de petits ARN interférents.
De plus, il est pratiquement important d’utiliser deux ARNSI ou plus ciblant des sites différents dans un gène cible visé, car les effets de knockdown d’un gène cible visé sont censés être communs, mais les effets hors cible sont susceptibles d’être différents entre les ARNSI.
Conclusion
L’efficacité de chaque ARNSI est connue pour être très variable en fonction de sa séquence dans les cellules de mammifères, et seule une fraction limitée des ARNSI conçus au hasard est fonctionnelle. De plus, des effets de silençage hors cible surviennent lorsque l’ARNsi présente une complémentarité partielle dans la région de la graine avec des gènes involontaires. Ici, sur la base de la machinerie de l’ARNi, nous avons décrit la conception rationnelle d’ARNSI fonctionnels à effet hors cible réduit, qui sont censés abattre un gène cible – spécifiquement.
Déclaration de conflit d’intérêts
Les auteurs déclarent que la recherche a été menée en l’absence de relations commerciales ou financières pouvant être interprétées comme un conflit d’intérêts potentiel.
Remerciements
Ce travail a été partiellement soutenu par des subventions du Ministère de l’Éducation, de la Culture, des Sports, de la Science et de la Technologie du Japon (MEXT) et du Projet d’Innovation Cellulaire (MEXT), ainsi que par un Projet de Recherche de base pour une Université privée; subvention du fonds de contrepartie à Kumiko Ui-Tei.
Amarzguioui, M., et Prydz, H. (2004). Un algorithme de sélection de séquences d’ARNSI fonctionnelles. Biochem. Biophys. Rés. Commun. 316, 1050–1058.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Birmingham, A., Anderson, E. M. Il y a un certain nombre de facteurs qui peuvent être pris en compte dans le calcul de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence de l’indice de référence. Les correspondances de semences de 3’UTR, mais pas l’identité globale, sont associées à des ARNi hors cibles. NAT. Méthodes 3, 199-204.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Castanotto, D., et Rossi, J. J. (2009). Les promesses et les pièges des thérapies basées sur l’interférence ARN. Nature 457, 426-433.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Chalk, A. M., et Sonnhammer, E. L. L. (2008). Recherche de spécificité d’ARNsi incorporant des données de tolérance de non-concordance. Bioinformatique 24, 1316-1317.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Cui, W., Ning, J., Naik, U. P. et Duncan, M. K. (2004). OptiRNAi et outil de conception d’ARNi. Comput. Méthodes Programmes Biomed. 75, 67–73.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Ding, Y., Chan, C. Y., et Lawrence, C.E. (2004). Serveur web Sfold pour le pliage statistique et la conception rationnelle des acides nucléiques. Acides nucléiques Res. 32, W135-W141.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K. et Tuschl, T. (2001). Les duplexes d’ARN à 21 nucléotides médient l’interférence de l’ARN dans les cellules de mammifères en culture. Nature 411, 494-498.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Elbashir, W. M., Harborth, J., Weber, K. et Tuschl, T. (2002). Analysis of gene function in somatic mammalian cells using small interfering RNA. Méthodes 26, 199-213.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit d’une espèce de poisson de la famille des Sauterelles. Base structurelle pour la reconnaissance spécifique à la base nucléotidique 5′ de l’ARN guide par l’homme AGO2. Nature 465, 818-822.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S., et Li, Tongbin. (2008). DDRM: et outil de conception siRNA en ligne efficace et généralement applicable. Bioinformatique 24, 2405-2406.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit de l’une des principales sources d’information sur la santé et la sécurité au travail. MicroRNA targeting specificity in mammals: determinants beyond seed pairing. Mol. Cellule 27, 91-105.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R. et Hannon, G. J. (2001). Argonaute 2 lien entre les analyses génétiques et biochimiques de l’ARNi. Science 293, 1146-1150.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Henschel, A., Buchholz, F., et Habermann, B. (2004). DEQOR : un outil web pour la conception et le contrôle de la qualité des SIRNA. Acides nucléiques Res. 32, W113-W120.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Horn, T., Sandmann, T., et Boutros, M. (2010). Conception et évaluation de bibliothèques à l’échelle du génome pour les écrans d’interférence d’ARN. Biol génomique. 11, R61.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit de l’un des plus importants ouvrages de référence de l’histoire de l’art et de l’histoire de l’Art. Le profilage d’expression révèle une régulation génique hors cible par l’ARNi. NAT. Biotechnol. 21, 635–637.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit de l’un des principaux ouvrages de référence de l’auteur. Silencing de transcription « hors cible » d’ARNsi répandu médié par la complémentarité des séquences de la région de la graine. ARN 12, 1179-1187.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Ketting, R. F. (2011). Les multiples visages de l’ARNi. Dev. Cellule 15, 148-161.
CrossRef Texte intégral
Il a été découvert le à l’Observatoire Astronomique de Moscou (2003). Les SIRNA et les MIARN fonctionnels présentent un biais de brin. Cellule 115, 209-216.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Ladunga, I. (2007). Un silençage génique plus complet par moins d’ARNSI: conception optimisée transparente et signature biophysique. Acides nucléiques Res. 35, 433-440.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Leuschner, P. J., Ameres, S. L., Kueng, S. et Martinez, J. (2006). Clivage du brin passager d’ARNsi lors de l’assemblage du RISC dans des cellules humaines. 7, 314-320.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit de l’une des principales sources d’information sur la santé et la santé au travail. L’appariement des graines conservées, souvent flanqué d’adénosines, indique que des milliers de gènes humains sont des cibles de microARN. Cellule 120, 15-20.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S. et Johnson, J. M. (2005). L’analyse de microréseaux montre que certains microARN régulent à la baisse un grand nombre d’ARNM cibles. Nature 433, 769-773.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Lu, J. J., et Mathews, D. H. (2008). Sélection efficace des ARNsi en utilisant la thermodynamique d’hybridation. Acide nucléique Res. 36, 640-647.
CrossRef Texte intégral
Il s’agit de l’un des principaux facteurs de croissance de la population. Base structurelle pour la reconnaissance spécifique de l’EXTRÉMITÉ 5′ de l’ARN guide par la protéine A. fulgidus piwi. Nature 434, 666-670.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R. et Tuschl, T. (2002). Les SIRNA antisens à simple brin guident le clivage de l’ARN cible dans l’ARNi. Cellule 110, 563-574.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., et Zamore, P.D. (2005). Le clivage passager-brin facilite l’assemblage des ARNsi en complexes enzymatiques ARNi contenant de l’Ago2. Cellule 123, 607-620.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Naito, Y., Yoshimura, J., Morishita, S. et Ui-Tei, K. (2009). siDirect 2.0: logiciel mis à jour pour la conception d’ARNSI fonctionnels avec un effet hors cible dépendant de la graine réduit. BMC Bioinformatique 10, 392.
CrossRef Texte intégral
Il s’agit d’une espèce de plantes de la famille des » Poaceae « , sous-famille des » Poaceae « , sous-famille des » pooideae « , originaire d’Asie du Sud-Est. AsiDesigner: serveur de conception siRNA basé sur un exon envisageant un épissage alternatif. Acide nucléique Res. 36, W97-W103.
CrossRef Texte intégral
Rand, T. A., Petersen, S., Du, F., et Wang, X. (2005). L’Argonaute2 clive le brin anti-guide de l’ARNSI lors de l’activation du RISC. Cellule 123, 621-629.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S. et Khvorova, A. (2004). Conception rationnelle de l’ARNSI pour l’interférence de l’ARN. NAT. Biotechnol. 22, 326–330.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit d’une espèce de plantes de la famille des » Poaceae « , sous-famille des » pooideae « , sous-famille des » Pooideae « , originaire d’Asie du Sud-Est. Asymétrie dans l’assemblage du complexe enzymatique de l’ARNi. Cellule 115, 199-208.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Shah, J. K., Garner, H. R., White, M. A., Shames, D. S., et Minna, J.D. (2007). sIR: siRNA information resource, un outil en ligne pour la conception et l’analyse des séquences d’ARNsi et une base de données d’ARNsi en libre accès. BMC Bioinformatique 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Texte Intégral
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A. et Saigo, K. (2008). La stabilité thermodynamique et l’appariement de la base Watoson-Crick dans le duplex de la graine sont des déterminants majeurs de l’efficacité de l’effet hors cible basé sur le sRNA. Acides nucléiques Res. 36, 7100-7109.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. et Saigo, K. (2004). Lignes directrices pour la sélection de séquences d’ARNSI hautement efficaces pour l’interférence d’ARN de mammifères et d’ARN de poussin. Acides nucléiques Res. 32, 936-948.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Vert, J.-P., Foveau, N., Lajaunie, C., et Vandenbrouck, Y. (2006). Un modèle précis et interprétable pour la prédiction de l’efficacité de l’ARNsi. BMC Bioinformatique 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Texte Intégral
Il s’agit d’une espèce de plantes de la famille des » Poaceae « , sous-famille des » pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « , sous-famille des » Pooideae « . Serveur de sélection siRAN : un serveur automatisé de prédiction d’oligonucléotides siRNA. Acides nucléiques Res. 32, W130-W134.
Résumé Pubmed / Texte Intégral Pubmed | Texte Intégral CrossRef
Il s’agit d’une espèce de poissons de la famille des Pygmées. La structure cristalline d’A. aeolicus argonaute, une endoribonucléase guidée par l’ADN spécifique à un site, fournit des informations sur le clivage de l’ARNm médié par le RISC. Mol. Cellule 19, 405-419.
Résumé Pubmed / Texte Intégral Pubmed / Texte Intégral CrossRef