- Introduction
- geoptimaliseerd ontwerp van siRNA
- stap 1: selectie van functionele Sirna-sequenties
- stap 2: Selectie van de siRNA-sequentie met verminderde Off-Target-Effecten
- stap 3: Eliminatie van bijna Perfect overeenkomende Off-Target genen
- siRNA ontwerpsoftware
- conclusie
- belangenverstrengeling verklaring
- Dankbetuigingen
Introduction
RNA interferentie (RNAi) is een breed gebruikte techniek waarbij klein interfererend RNA (siRNA) een specifiek doelgen met een perfecte complementaire sequentie downreguleert, en beloofde te gebruiken in therapeutische toepassingen voor ziekten bij de mens (Castanotto and Rossi, 2009; Ketting, 2011). Hoewel de mens meer dan 20.000 genen heeft, is het wenselijk om siRNA te gebruiken die hoogst functioneel is en geen gevolgen op om het even welke genen buiten zijn specifiek doel heeft. In dit artikel bekijken we een geoptimaliseerde methode om siRNA te ontwerpen op basis van het mechanisme van RNAi. Daarnaast introduceren we de websites die toegankelijk zijn voor het publiek voor het selecteren van siRNA-sequenties.
geoptimaliseerd ontwerp van siRNA
duplexen van 21-nucleotide (nt) RNA met 2 NT 3′ overhangen (siRNA) wordt gewoonlijk gebruikt voor RNAi-experimenten. Bij levering in de cellen, wordt siRNAs opgenomen in RNA-veroorzaakte complex tot zwijgen brengen (RISC) als double-stranded RNA. RISC is het effectorcomplex dat Argonaute-eiwit (Ago) bevat met sliceractiviteit (Hammond et al., 2001; Martinez et al., 2002). De siRNA-geleidingsstreng met het thermodynamisch minder stabiele 5′ – uiteinde wordt bij voorkeur vastgehouden door RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). De passagiersstrengen van de meeste dubbelstrengs siRNAs die op RISC worden geladen worden gespleten door Ago2 proteã ne en gedegradeerd (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). De behouden paren van de gidsband richten mRNA met volkomen complementaire opeenvolging, en onderdrukt het door splitsing door ago2 proteã ne bij nucleotidepositie 10 van siRNA gidsband (Elbashir et al., 2001; Hammond et al., 2001; Martínez et al., 2002). Nochtans, wijst een geaccumuleerd bewijsmateriaal van genoom-brede experimenten erop dat een groot aantal mRNAs met gedeeltelijke complementariteit aan de gidsband ook worden verminderd (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Dit fenomeen wordt aangeduid als zaad-afhankelijk off-target effect en bij voorkeur waargenomen in mRNA 3 ‘ Utr ‘ s. Het doelherkenningsmechanisme van dit off-target effect is gekend gelijkaardig aan dat van miRNA-gemedieerde gen het tot zwijgen brengen (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). De afschriften met opeenvolgingen complementair aan het zaadgebied geplaatst 2-8 van de 5’ terminal worden hoofdzakelijk verminderd. Het zaadgebied is bekend te zijn gelegen op het oppervlak van Ago in een quasi-spiraalvormige vorm te dienen als de ingang of nucleatie site voor kleine RNAs in de RISCs (Ma et al., 2005; Yuan et al., 2005). Aldus, identificeert het zaadgebied eerst het doel mRNAs, en vormt vervolgens het perfecte basis-in paren rangschikken met voorgenomen doel mRNA en induceert RNAi door Ago2.
op basis van het mechanisme van RNAi wordt een genspecifieke siRNA als selecteerbaar beschouwd volgens de volgende drie stappen.
stap 1: selectie van functionele Sirna-sequenties
de neerslagefficiëntie van siRNA ‘ s blijkt sterk afhankelijk te zijn van hun sequenties. We pleitten voor de empirisch gebaseerde regel die de kenmerken van zeer functionele siRNAs voorgeschreven (Ui-Tei et al., 2004), een dergelijke regel wordt genoemd als Ui-Tei regel (figuur 1). De door Ui-Tei-regel gekozen siRNA voldoet tegelijkertijd aan de volgende vier voorwaarden:: (1) A of U op positie 1 vanaf 5′ eindpunt van siRNA-geleidingsstreng, (2) G of C op positie 19, (3) au-rijkheid (AU ≥4) op posities 1-7, en (4) geen lange GC-rek ≥10. Met uitzondering van (4), gaf onze regel aan dat de functionele siRNA asymmetrische stabiliteit heeft in 5′ en 3′ terminals. Onze experimentele validatie met behulp van luciferase reporter assay toonde aan dat 98% van de siRNAs voldoen aan bovenstaande voorwaarden verminderde de expressie van luciferase reporter onder 33% (Ui-Tei et al., 2004). Andere groepen demonstreerden ook de regels van zeer functionele siRNAs aangeduid als Reynolds regel (Reynolds et al., 2004) en amarzguioui rule (Amarzguioui en Prydz, 2004) zoals samengevat in Figuur 1. Deze regels toonden ook duidelijk aan dat functionele siRNAs asymmetrisch zijn: een bundel van RNA met onstabiele 5’ terminal was efficiënt als gidsbundel. Bovendien, gemeenschappelijk in deze regels, 5 ‘ terminus van functionele siRNA gids streng was de voorkeur aan A of U. het werd later onthuld dat de resultaten weerspiegeld de structurele kenmerken van de menselijke Ago2 (Frank et al., 2010). De kristalstructuur van een Midden (Midden) domein van menselijke ago2 en NMR titratieexperimenten toonde aan dat nucleotidemonofosfaten, AMP en UMP, met tot 30-voudige hogere affiniteit dan of CMP of GMP binden, die structureel bewijsmateriaal voor nucleotide-specifieke interactie in het MIDDENDOMEIN van eukaryotic geleden proteã nen verstrekken.
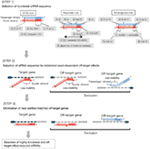
figuur 1. Schematische weergave voor het selecteren van functionele en off-target effect-gereduceerde siRNAs. Selectie van zeer functionele siRNA door Ui – Tei, Reynolds, amarzguioui regels, of de combinatie van hen (stap 1). Selectie van siRNAs met lage stabiliteit in de seed-target duplexen (stap 2). Eliminatie van siRNAs met bijna perfect overeenkomende sequenties aan niet-doelgenen (stap 3). In elke regel, geeft de nucleotidepositie het aantal nucleotide aan dat van 5′ eind van de geleidingsbundel wordt geteld.  :
:  A / U op positie 1.
A / U op positie 1.  G / C op positie 19.
G / C op positie 19.  4 tot 7 A / V in posities 1-7.
4 tot 7 A / V in posities 1-7. 
 :
:  GC-gehalte (30% -52%).
GC-gehalte (30% -52%).  A / U ≥ 3 op posities 1-5.
A / U ≥ 3 op posities 1-5.  afwezigheid van interne herhalingen.
afwezigheid van interne herhalingen.  A op positie 1.
A op positie 1.  A op positie 17.
A op positie 17.  u op positie 10.
u op positie 10.  niet G / C op positie 1.
niet G / C op positie 1.  niet G op positie 7.
niet G op positie 7. 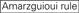 :
:  een asymmetrie in de stabiliteit van de duplex-einden (gemeten als het A/U-differentieel van de drie terminalbasepairs aan beide uiteinden van de duplex).
een asymmetrie in de stabiliteit van de duplex-einden (gemeten als het A/U-differentieel van de drie terminalbasepairs aan beide uiteinden van de duplex).  G of C op positie 19.
G of C op positie 19.  A of U op positie 1.
A of U op positie 1.  A of U op positie 14.
A of U op positie 14.  niet U op positie 19.
niet U op positie 19.  niet G op positie 1.
niet G op positie 1.
stap 2: Selectie van de siRNA-sequentie met verminderde Off-Target-Effecten
om zaadafhankelijke off-target-effecten te vermijden, kan een aanpak zijn om de siRNA-guide-streng te selecteren waarvan de seed-sequentie niet complementair is aan alle sequenties in de 3′ UTR van alle niet-gerichte genen. Nochtans, is deze benadering gebleken onmogelijk te zijn, omdat menselijke siRNAs met de meest zeldzame Seven-nt zaadvolgorde nog zaad-complementariteit met verscheidene niet-gerichte mRNAs had. Zo, hebben we gezocht naar de regels die het vermogen van siRNAs regelen om zaad-afhankelijk off-target effect te veroorzaken, en onthulde dat de efficiëntie van off-target effect is sterk gecorreleerd aan de thermodynamische stabiliteit van de duplex gevormd tussen het zaadgebied van siRNA gids streng en zijn doel mRNA (Ui-Tei et al., 2008). De smelttemperatuur (Tm), een van de thermodynamische parameters voor de vorming van RNA-duplex, toonde een sterke positieve correlatie met de inductie van zaadafhankelijke off-target effecten. Dus, het selecteren van de siRNAs met lage Tm van de seed-target duplex moet zaad-afhankelijke off-target silencing minimaliseren (figuur 1). De Tm van 21,5°C kan als benchmark dienen, waardoor de bijna off-target-vrije zaadsequenties worden onderscheiden van de off-target-positieve. Aangezien het off-target effect niet alleen kan worden veroorzaakt door de geleidingsstreng, maar ook door de passagiersstreng, zijn siRNAs waarvan de seed-target Tm voldoende laag is voor beide strengen gunstig.
stap 3: Eliminatie van bijna Perfect overeenkomende Off-Target genen
zelfs wanneer de Tm-waarde van de seed-target duplex voldoende laag is, kan het tot zwijgen brengen van het doelgen nog steeds plaatsvinden als het niet-zaadgebied volledig complementair is. Daarom, in de derde stap, siRNAs die bijna perfecte overeenkomsten met een andere niet-gerichte transcripten zijn geëlimineerd (figuur 1).
siRNA ontwerpsoftware
we presenteerden siRNA ontwerpsoftware, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figuur 2), die functionele, doelspecifieke siRNA-ontwerpsoftware biedt volgens de hierboven genoemde procedures (Naito et al., 2009). In standaard parameter, siRNAs voldoen aan Ui-Tei regel kan worden geselecteerd. Wanneer de kandidaat functionele siRNA ’s zaad-doel duplexen met Tm-waarden onder 21,5°C kunnen vormen, en hun 19-nt regio’ s die posities 2-20 van beide strengen overspannen ten minste twee mismatches hebben met andere niet-gerichte transcripten, kan siDirect 2.0 ten minste één gekwalificeerde siRNA ontwerpen voor >94% van de menselijke mRNA-sequenties in RefSeq.

Figuur 2. Schermweergaven van siDirect 2.0 siRNA ontwerpsoftware. (A) bovenste bladzijde (http://siDirect2.RNAi.jp/). B) facultatieve parameters voor het ontwerp van siRNA. (C) resultaatpagina. (D) gedetailleerde lijst van off-target kandidaten met bijna perfecte matches. De uitlijning tussen elk off-target transcript en de siRNA sequentie visualiseert de posities van mismatches.
andere software voor het selecteren van functionele siRNAs waren open voor het publiek, zoals weergegeven in Tabel 1. In veel van deze, Ui-Tei regel (Ui-Tei et al., 2004), Reynolds rule (Reynolds et al., 2004), amarzguioui rule (amarzguioui en Prydz, 2004), Tuschl rule (Elbashir et al., 2002), en de combinatie van hen werden vaak en op grote schaal gebruikt. Om de bijna perfecte overeenkomende niet-doelgenen te elimineren, werd BLAST search gebruikt voor homologieonderzoek in verschillende software. Echter, omdat BLAST search is niet zo nauwkeurig voor korte sequenties zoals siRNAs, siDirect, WU-BLAST, en Bowtie, die zeer nauwkeurige homologie zoekmachine voor korte sequenties worden vaak gebruikt. Onder hen, siDirect 2.0 kan de meest nauwkeurige resultaten te brengen. Bovendien, sommige van de software overwegen de extra functies, zoals mRNA secundaire structuur (Ladunga, 2007; Lu en Mathews, 2008), alternatieve splicing (Park et al., 2008), of de motiefvolgorde waarmee de immuunrespons door een RNA-virus kan optreden (Gong et al., 2008). Deze functies worden niet beschouwd in siDirect 2.0. Dus, om rekening te houden met deze functies, siRNAs vaak geselecteerd door siDirect 2.0 en de andere geschikte software programma ‘ s kunnen een optimaal resultaat te produceren.
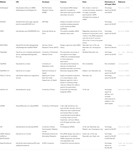
Tabel 1. Kleine interfererende RNA ontwerpsoftware programma ‘ s.
bovendien is het praktisch belangrijk om twee of meer siRNAs te gebruiken die zich richten op verschillende plaatsen in een beoogd doelgen, aangezien de “knockdown” -effecten van een beoogd doelgen algemeen worden geacht, maar de “off-target” – effecten waarschijnlijk verschillen tussen siRNAs.
conclusie
het is bekend dat de werkzaamheid van elk siRNA sterk varieert, afhankelijk van de sequentie in zoogdiercellen, en slechts een beperkte fractie van willekeurig ontworpen siRNA ‘ s is functioneel. Bovendien doen zich off-target het tot zwijgen brengen gevolgen voor wanneer siRNA gedeeltelijke complementariteit in het zaadgebied met onbedoelde genen heeft. Hier, gebaseerd op de RNAi-machines, beschreven we het rationele ontwerp van functionele, off-target effect-verminderde siRNAs, die naar verwachting een doelgen-specifiek neerhalen.
belangenverstrengeling verklaring
de auteurs verklaren dat het onderzoek werd uitgevoerd in afwezigheid van commerciële of financiële relaties die als een potentieel belangenconflict kunnen worden opgevat.
Dankbetuigingen
dit werk werd gedeeltelijk ondersteund door subsidies van het Ministerie van Onderwijs, Cultuur, Sport, Wetenschap en technologie van Japan (MEXT), en het Cell Innovation Project (MEXT), en Kernonderzoek voor particuliere universiteit; matching fund subsidie aan Kumiko Ui-Tei.
Amarzguioui, M., en Prydz, H. (2004). Een algoritme voor de selectie van functionele siRNA-sequenties. Biochem. Biophys. Res. Commun. 316, 1050–1058.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S., and Khvorova, A. (2006). 3 ‘ UTR seed matches, maar niet de totale identiteit, worden geassocieerd met RNAi off-targets. Nat. Methods 3, 199-204.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Castanotto, D., and Rossi, J. J. (2009). De beloften en valkuilen van RNA-interferentie-gebaseerde therapieën. Natuur 457, 426-433.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Chalk, A. M., and Sonnhammer, E. L. L. (2008). siRNA specificiteit zoeken inclusief mismatch tolerantiegegevens. Bioinformatica 24, 1316-1317.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Cui, W., Ning, J., Naik, U. P., and Duncan, M. K. (2004). OptiRNAi, en RNAi ontwerp tool. Berekenen. Methoden Programma ‘ S Biomed. 75, 67–73.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Ding, Y., Chan, C. Y., and Lawrence, C. E. (2004). Sfold web server voor statistische vouwen en rationeel ontwerp van nucleïnezuren. Nucleïnezuren Res. 32, W135-W141.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K., and Tuschl, T. (2001). Duplexen van 21-nucleotide RNAs bemiddelen RNA interferentie in gekweekte zoogdiercellen. Nature 411, 494-498.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Elbashir, W. M., Harborth, J., Weber, K., and Tuschl, T. (2002). Analyse van genfunctie in somatische zoogdiercellen met behulp van kleine interfererende RNAs. Methods 26, 199-213.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Frank, F., Sonenberg, N., en Nagar, B. (2010). Structurele basis voor 5 ‘ – nucleotide base-specifieke erkenning van gidsrna door menselijk AGO2. Nature 465, 818-822.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S., en Li, Tongbin. (2008). siDRM: en effectieve en algemeen toepasbare online siRNA design tool. Bioinformatica 24, 2405-2406.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P., and Bartel, D. P. (2007). MicroRNA targeting specificity in mammals: determinants beyond seed pairing. Mol. Cel 27, 91-105.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R., and Hannon, G. J. (2001). Argonaute 2 link tussen genetische en biochemische analyses van RNAi. Wetenschap 293, 1146-1150.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Henschel, A., Buchholz, F., en Habermann, B. (2004). DEQOR: een web-based tool voor het ontwerp en de kwaliteitscontrole van siRNAs. Nucleïnezuren Res. 32, W113-W120.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Horn, T., Sandmann, T., and Boutros, M. (2010). Ontwerp en evaluatie van genoombrede bibliotheken voor RNA-interferentieschermen. Genome Biol. 11, R61.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Jackson, A. L., Bartz, S. R., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., and Linsley, P. S. (2003). Expressie profilering onthult off-target gen regelgeving door RNAi. Nat. Biotechnol. 21, 635–637.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L., and Linsley, P. S. (2006). Wijdverspreide siRNA” off-target ” transcript silencing gemedieerd door zaad regio sequentie complementariteit. RNA 12, 1179-1187.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
(2011). De vele gezichten van RNAi. Dev. Cel 15, 148-161.
CrossRef volledige tekst
Khvorova, A., Reynolds, A., and Jayasena, S. D. (2003). Functionele siRNAs en miRNAs vertonen bundel bias. Cel 115, 209-216.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Ladunga, I. (2007). Meer volledige gen het tot zwijgen brengen door minder siRNAs: transparant geoptimaliseerd ontwerp en Biofysische handtekening. Nucleïnezuren Res. 35, 433-440.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Leuschner, P. J., Ameres, S. L., Kueng, S., and Martinez, J. (2006). Splitsing van de siRNA passenger strand tijdens RISC assemblage in menselijke cellen. EMBO Rep. 7, 314-320.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Lewis, B. P., Burge, C. B., and Bartel, D. P. (2005). Het behouden zaad in paren rangschikken, vaak geflankeerd door adenosines, wijst erop dat duizenden menselijke genen microRNA-doelwitten zijn. Cel 120, 15-20.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S., and Johnson, J. M. (2005). De microarray analyse toont aan dat sommige microRNAs grote aantallen doelmrnas downreguleren. Nature 433, 769-773.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Lu, Z. J., and Mathews, D. H. (2008). Efficiënte siRNA selectie met behulp van hybridisatie thermodynamica. Nucleïnezuur Res. 36, 640-647.
CrossRef volledige tekst
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T., and Patel, D. J. (2005). Structurele basis voor 5 ‘ – end – specifieke herkenning van guide RNA door het A. fulgidus piwi eiwit. Nature 434, 666-670.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R., and Tuschl, T. (2002). Single-stranded antisense siRNAs gidsdoelrna splitsing in RNAi. Cel 110, 563-574.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., and Zamore, P. D. (2005). Passenger-strand splitsing vergemakkelijkt assemblage van siRNA in Ago2-bevattende RNAi enzym complexen. Cel 123, 607-620.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Naito, Y., Yoshimura, J., Morishita, S., en Ui-Tei, K. (2009). siDirect 2.0: bijgewerkte software voor het ontwerpen van functionele siRNA met verminderd zaad-afhankelijk off-target effect. BMC Bioinformatics 10, 392.
CrossRef volledige tekst
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M., en Kim, Y. J. (2008). AsiDesigner: exon-gebaseerde siRNA design server overweegt alternatieve splicing. Nucleïnezuur Res. 36, W97-W103.
CrossRef volledige tekst
Rand, T. A., Petersen, S., Du, F., and Wang, X. (2005). Argonaute2 splijt de anti-guide streng van siRNA tijdens RISC activering. Cel 123, 621-629.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S., and Khvorova, A. (2004). Rationeel siRNA-ontwerp voor RNA-interferentie. Nat. Biotechnol. 22, 326–330.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N., and Zamore, P. D. (2003). Asymmetrie in de assemblage van het RNAi enzym complex. Cel 115, 199-208.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Shah, J. K., Garner, H. R., White, M. A., Shames, D. S., and Minna, J. D. (2007). siRNA information resource, een web-based tool voor siRNA sequence design en analyse en een open access siRNA database. BMC Bioinformatics 8, 178. doi:10.1186/1471-2105-8-178
volledige tekst CrossRef
Ui – Tei, K., Naito, Y., Nishi, K., Juni, A., en Saigo, K. (2008). Thermodynamische stabiliteit en watoson-Crick base pairing in de zaad duplex zijn belangrijke determinanten van de efficiëntie van de sRNA-gebaseerde off-target effect. Nucleïnezuren Res. 36, 7100-7109.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Ui – Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. en Saigo, K. (2004). Richtlijnen voor de selectie van zeer efficiënte siRNA-sequenties voor de interferentie van RNA van zoogdieren en kuikens. Nucleïnezuren Res. 32, 936-948.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Vert, J.-P., Foveau, N., Lajaunie, C., en Vandenbrouck, Y. (2006). Een accuraat en interpreteerbaar model voor siRNA-werkzaamheidsvoorspelling. BMC Bioinformatics 7, 520. doi:10.1186/1471-2105-7-520
volledige tekst CrossRef
Yuan, B., Latek, R., Rossbach, M., Tuschl, T., and Lewitter, F. (2004). siRAN selection server: een geautomatiseerde siRNA oligonucleotide voorspelling server. Nucleïnezuren Res. 32, W130–W134.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T., and Patel, D. J. (2005). De kristalstructuur van A. aeolicus argonaute, een plaats-specifieke DNA-geleide endoribonuclease, verstrekt inzicht in RISC-gemedieerde mRNA splitsing. Mol. Cel 19, 405-419.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text