introduktion
RNA-interferens (RNAi) er en bredt anvendt teknik, hvorved lille interfererende RNA (siRNA) nedregulerer et specifikt målgen med perfekt komplementær sekvens og lovede at bruge i terapeutisk anvendelse til humane sygdomme (Castanotto og Rossi, 2009; Ketting, 2011). Selvom mennesket har mere end 20.000 gener, er det ønskeligt at anvende siRNA, som er yderst funktionel og ikke har nogen virkning på andre gener end dets specifikke mål. I denne artikel gennemgår vi en optimeret metode til at designe siRNA baseret på RNAi-mekanismen. Derudover introducerer vi de hjemmesider, der er åbne for offentligheden til valg af siRNA-sekvenser.
optimeret Design af siRNA
duplekser af 21-nukleotid (nt) RNA med 2 NT 3′ overhæng (siRNA) bruges normalt til RNAi-eksperimenter. Ved levering til cellerne inkorporeres sirna ‘ er i det RNA-inducerede lyddæmpningskompleks (RISC) som et dobbeltstrenget RNA. RISC er effektorkomplekset indeholdende Argonaute protein (Ago) med skæreaktivitet (Hammond et al., 2001; Martinus et al., 2002). SiRNA-styrestrengen indeholdende den termodynamisk mindre stabile 5 ‘ – ende bevares fortrinsvis af RISC (Khvorova et al., 2003; et al., 2003; Ui-Tei et al., 2004). Passagerstrengene i de fleste af de dobbeltstrengede sirna ‘ er, der er indlæst på RISC, spaltes af Ago2-protein og nedbrydes (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). De tilbageholdte styrestrengspar retter sig mod mRNA med perfekt komplementær sekvens og undertrykker det ved spaltning af Ago2-protein ved nukleotidposition 10 af siRNA-styrestreng (Elbashir et al., 2001; Hammond et al., 2001; Martinus et al., 2002). Imidlertid indikerer et akkumuleret bevis fra genomdækkende eksperimenter, at et stort antal mRNA ‘ er med delvise komplementariteter til styrestrengen også reduceres (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Dette fænomen kaldes frøafhængig off-target-effekt og observeres fortrinsvis i mRNA 3’ UTR ‘ er. Målgenkendelsesmekanismen for denne off-target-effekt er kendt for at være den samme som for miRNA-medieret genhæmning., 2005; Lim et al., 2005; Grimson et al., 2007). Transkriptionerne med sekvenser, der supplerer frøområdet placeret 2-8 fra 5’ – terminalen, reduceres hovedsageligt. Frøområdet er kendt for at være placeret på overfladen af Ago i en kvasi-spiralformet form for at tjene som indgangs-eller nukleationsstedet for små RNA ‘ er i Risics (Ma et al., 2005; Yuan et al., 2005). Således identificerer frøområdet først mål-mRNA ‘ erne og danner derefter perfekt baseparring med det tilsigtede mål-mRNA og inducerer RNAi af Ago2.
baseret på mekanismen for RNAi anses et målgenspecifikt siRNA for at være valgbart i henhold til de følgende tre trin.
trin 1: Valg af funktionel siRNA-sekvens
nedbrydningseffektiviteten af siRNA ‘ er afsløres at være meget afhængig af deres sekvenser. Vi foreslog den empirisk baserede regel, der foreskrev egenskaberne ved meget funktionelle sirna ‘ er (Ui-Tei et al., 2004), kaldes en sådan regel som Ui-Tei-regel (Figur 1). SiRNA valgt af Ui-Tei-reglen opfylder følgende fire betingelser samtidigt: (1) A eller U i position 1 fra 5′ endestation af siRNA guide streng, (2) G eller C i position 19, (3) AU rigdom (AU kr4) i position 1-7, og (4) ingen lang GC strække kr10. Bortset fra (4) angav vores regel, at det funktionelle siRNA har asymmetrisk stabilitet i 5′ og 3’ terminaler. Vores eksperimentelle validering ved hjælp af luciferase reporter assay viste, at 98% af siRNA ‘ erne, der opfyldte ovenstående betingelser, reducerede ekspressionen af luciferase reporter under 33% (Ui-Tei et al., 2004). Andre grupper demonstrerede også reglerne for meget funktionelle sirna ‘ er kaldet Reynolds rule (Reynolds et al., 2004) og Amarguioui-reglen (Amarguioui og Pryds, 2004) som opsummeret i Figur 1. Disse regler viste også tydeligt, at funktionelle sirna’ er er asymmetriske: en RNA-streng med ustabil 5 ‘ – terminal var effektiv som en styrestreng. Desuden, almindelig i disse regler, 5’ terminal af funktionel siRNA guide streng var at foretrække at være A eller U. det blev afsløret senere, at resultaterne afspejlede de strukturelle træk ved human Ago2 (Frank et al., 2010). Krystalstrukturen i et midt (mellem) domæne fra humane AGO2-og NMR-titreringseksperimenter viste, at nukleotidmonophosphater, AMP og UMP, binder med op til 30 gange højere affinitet end enten CMP eller GMP, hvilket giver strukturelle beviser for nukleotidspecifikke interaktioner i MELLEMDOMÆNET for eukaryote ago-proteiner.
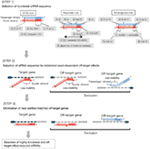
Figur 1. Skematisk repræsentation til valg af funktionelle og off-target effekt-reducerede sirna ‘ er. Udvælgelse af yderst funktionel siRNA ved Ui-Tei, Reynolds, Amarguioui regler, eller kombinationen af dem (trin 1). Udvælgelse af siRNA ‘ er med lav stabilitet i frømålduplekserne (trin 2). Eliminering af siRNA ‘ er med næsten perfekte matchede sekvenser til ikke-målgener (trin 3). I hver regel angiver nukleotidpositionen antallet af nukleotid talt fra 5 ‘ terminal af styrestrengen.  :
:  A/U ved position 1.
A/U ved position 1.  G / C ved position 19.
G / C ved position 19.  4 til 7 A/Us i position 1-7.
4 til 7 A/Us i position 1-7. 
 :
:  GC-indhold (30% -52%).
GC-indhold (30% -52%).  a / u 3 i position 1-5.
a / u 3 i position 1-5.  fravær af interne gentagelser.
fravær af interne gentagelser.  A i position 1.
A i position 1.  A ved position 17.
A ved position 17.  U i position 10.
U i position 10.  ikke G/C i position 1.
ikke G/C i position 1.  ikke G i position 7.
ikke G i position 7. 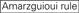 :
:  en asymmetri i stabiliteten af dupleksenderne (målt som A/U-differensen for de tre terminale basepar i hver ende af dupleksen).
en asymmetri i stabiliteten af dupleksenderne (målt som A/U-differensen for de tre terminale basepar i hver ende af dupleksen).  G eller C i position 19.
G eller C i position 19.  A eller U i position 1.
A eller U i position 1.  A eller U i position 14.
A eller U i position 14.  ikke U i position 19.
ikke U i position 19.  ikke G i position 1.
ikke G i position 1.
trin 2: Udvælgelse af siRNA-sekvens med reducerede off-Target-effekter
for at undgå frøafhængige off-target-effekter kan en tilgang være at vælge siRNA-styrestrengen, hvis frøsekvens ikke er komplementær til nogen sekvenser i 3′ UTR for alle ikke-målrettede gener. Imidlertid har denne tilgang vist sig at være umulig, fordi menneskelige sirna ‘er med den mest sjældne syv-nt-frøsekvens stadig havde frøkomplementariteter med flere ikke-målrettede mRNA’ er. Så vi har kigget efter de regler, der styrer siRNA ‘ ers evne til at inducere frøafhængig off-target-effekt, og afslørede, at effektiviteten af off-target-effekt er stærkt korreleret med den termodynamiske stabilitet af dupleksen dannet mellem frøregionen af siRNA guide streng og dens mål mRNA (Ui-Tei et al., 2008). Smeltetemperaturen (Tm), en af de termodynamiske parametre til dannelse af RNA-dupleks, viste stærk positiv korrelation med induktionen af frøafhængige off-target-effekter. Således bør valg af siRNA ‘ er med lav Tm for frømål-dupleksen minimere frøafhængig lyddæmpning uden for målet (Figur 1). Tm på 21,5 kr. C kan tjene som benchmark, som diskriminerer de næsten off-target-fri frøsekvenser fra de off-target-positive. Da off-target-effekten desuden kan være forårsaget af ikke kun af føringsstrengen, men også af passagerstrengen, er sirna ‘ er, hvis seed-target Tm er tilstrækkelig lav til begge tråde, gunstige.
trin 3: Eliminering af næsten perfekte matchede off-Target-gener
selv når TM-værdien af frø-target-dupleksen er tilstrækkelig lav, kan målgenhæmningen stadig finde sted, hvis ikke-frøområdet er fuldstændigt komplementært. I det tredje trin blev sirna ‘ er, der har næsten perfekte match til andre ikke-målrettede udskrifter, derfor elimineret (Figur 1).
Sirna designprogram
vi præsenterede siRNA designprogram, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figur 2), som tilvejebringer funktionelt, målspecifikt siRNA-designprogram i henhold til ovennævnte procedurer (Naito et al., 2009). I standardparameter kan siRNAs tilfredsstillende Ui-Tei-regel vælges. Når kandidatfunktionelle sirna ‘ er kunne danne Seed-target-duplekser med Tm-værdier under 21,5 C, og deres 19-nt-regioner, der spænder over positioner 2-20 af begge tråde, har mindst to uoverensstemmelser med andre ikke-målrettede udskrifter, kan siDirect 2.0 designe mindst et kvalificeret siRNA til >94% af humane mRNA-sekvenser i Refseks.

figur 2. Skærmvisninger af siDirect 2.0 siRNA designprogram. (A) øverste side (http://siDirect2.RNAi.jp/). B) valgfrie parametre for siRNA-design. (C) resultat side. (D) detaljeret liste over kandidater uden for målet med næsten perfekte kampe. Justeringen mellem hvert off-target-transkript og siRNA-sekvensen visualiserer positionerne for uoverensstemmelser.
andre programmer til valg af funktionelle sirna ‘ er var åbne for offentligheden som vist i tabel 1. I mange af dem, Ui-Tei regel (Ui-Tei et al., 2004), Reynolds rule (Reynolds et al., 2004), Amarguioui-reglen (Amarguioui og Pryds, 2004), Tuschl-reglen (Elbashir et al., 2002), og kombinationen af dem blev ofte og meget udbredt. For at eliminere de næsten perfekte matchede ikke-målgener blev BLAST search brugt til homologisøgning i flere programmer. Men da BLAST søgning ikke er så præcis for korte sekvenser som sirna ‘ er, siDirect, VU-BLAST og Butterfly, som er meget nøjagtige homologi søgemaskine til korte sekvenser bruges ofte. Blandt dem kan siDirect 2.0 give de mest nøjagtige resultater. Desuden overvejer nogle af programmerne de ekstra funktioner, såsom mRNA sekundær struktur (Ladunga, 2007; Lu og matematik, 2008), alternativ splejsning (Park et al., 2008) eller motivsekvensen, hvormed immunresponset af en RNA-virus kan forekomme (Gong et al., 2008). Disse funktioner betragtes ikke i siDirect 2.0. Så for at tage højde for disse funktioner kan sirna ‘ er, der almindeligvis vælges af siDirect 2.0 og de andre passende programmer, give et optimalt resultat.
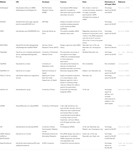
tabel 1. Små interfererende RNA design programmer.
derudover er det praktisk taget vigtigt at overveje at bruge to eller flere sirna ‘er, der er målrettet mod forskellige steder i et tilsigtet målgen, da nedslagseffekterne af et tilsigtet målgen formodes at være almindelige, men off-target-effekterne sandsynligvis vil være forskellige mellem sirna’ er.
konklusion
effekten af hvert siRNA er kendt for at variere meget afhængigt af dets sekvens i pattedyrceller, og kun en begrænset brøkdel af tilfældigt designede sirna ‘ er er funktionel. Desuden opstår off-target lyddæmpningseffekter, når siRNA har delvis komplementaritet i frøområdet med utilsigtede gener. Her, baseret på RNAi-maskineriet, beskrev vi det rationelle design af funktionelle, off-target effekt-reducerede sirna ‘ er, som forventes at slå et målgen ned-specifikt.
interessekonflikt Erklæring
forfatterne erklærer, at forskningen blev udført i mangel af kommercielle eller økonomiske forhold, der kunne fortolkes som en potentiel interessekonflikt.
anerkendelser
dette arbejde blev delvist støttet af tilskud fra Japans ministerium for Uddannelse, Kultur, Sport, Videnskab og teknologi (MEKST) og Cell Innovation Project (MEKST) og Kerneforskningsprojekt for privat universitet; matchende fondstilskud til Kumiko Ui-Tei.
Amarguioui, M., Og Pryds, H. (2004). En algoritme til udvælgelse af funktionelle siRNA-sekvenser. Biochem. Biofys. Res. Commun. 316, 1050–1058.
Pubmed Abstrakt / Pubmed Fuld Tekst / CrossRef Fuld Tekst
Birmingham, A., Anderson, E. M. D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilov, J., Marshall, V. S. og Khvorova, A. (2006). 3 ‘ UTR seed kampe, men ikke samlet identitet, er forbundet med RNAi off-mål. Nat. Metoder 3, 199-204.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Castanotto, D., og Rossi, J. J. (2009). Løfterne og faldgruberne ved RNA-interferensbaseret terapi. Natur 457, 426-433.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Chalk, A. M. Og Sonnhammer, E. L. L. (2008). siRNA specificitet søgning inkorporerer mismatch tolerance data. Bioinformatik 24, 1316-1317.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
cui, ning, J., Naik, U. P. og Duncan, M. K. (2004). OptiRNAi, og RNAi design værktøj. Comput. Metoder Programmer Biomed. 75, 67–73.
Pubmed Abstrakt / Pubmed Fuld Tekst / CrossRef Fuld Tekst
Ding, Y., Chan, C. Y.(2004). Sfold server til statistisk foldning og rationel design af nukleinsyrer. Nukleinsyrer Res. 32, 135–141.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Elbashir, S. M., Harborth, J., Lendeckel, V., Yalcin, A., Viber, K. og Tuschl, T. (2001). Duplekser af 21-nukleotid-RNA ‘ er medierer RNA-interferens i dyrkede pattedyrceller. Natur 411, 494-498.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Elbashir, Harborth, J., K. og Tuschl, T. (2002). Analyse af genfunktion i somatiske pattedyrceller ved anvendelse af små interfererende RNA ‘ er. Metoder 26, 199-213.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Frank, F., Sonenberg, N. og Nagar, B. (2010). Strukturelt grundlag for 5 ‘ – nukleotidbasespecifik genkendelse af guide RNA af human AGO2. Natur 465, 818-822.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
det er en af de mest populære måder at gøre det på. (2008). siDRM: og effektiv og generelt anvendelig online siRNA design værktøj. Bioinformatik 24, 2405-2406.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Grimson, A., Farh, K. K., Johnston, Garrett-Engele, P., Lim, L. P. og Bartel, D. P. (2007). MicroRNA målrettet specificitet hos pattedyr: determinanter ud over frøparring. Mol. Celle 27, 91-105.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R. og Hannon, G. J. (2001). Argonaute 2 forbindelse mellem genetiske og biokemiske analyser af RNAi. Videnskab 293, 1146-1150.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Henschel, A., Buchholts, F. Og Habermann, B. (2004). Et internetbaseret værktøj til design og kvalitetskontrol af siRNA ‘ er. Nukleinsyrer Res. 32, 113–120.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Horn, T., Sandmann, T. og Boutros, M. (2010). Design og evaluering af genom-dækkende biblioteker til RNA-interferensskærme. Genom Biol. 11, R61.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Jackson, A. L., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G. og Linsley, P. S. (2003). Ekspressionsprofilering afslører genregulering uden for målet af RNAi. Nat. Biotechnol. 21, 635–637.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L. og Linsley, P. S. (2006). Udbredt siRNA” off-target ” transkript silencing medieret af frø region sekvens komplementaritet. RNA 12, 1179-1187.
Pubmed Abstrakt / Pubmed Fuld Tekst / CrossRef Fuld Tekst
Ketting, R. F. (2011). Rnais mange ansigter. Dev. Celle 15, 148-161.
CrossRef Fuld tekst
Khvorova, A., Reynolds, A. og Jayasena, S. D. (2003). Funktionelle sirna ‘er og miRNA’ er udviser strand bias. Celle 115, 209-216.
Pubmed Abstrakt / Pubmed Fuld Tekst / CrossRef Fuld Tekst
Ladunga, I. (2007). Mere komplet genhæmning af færre sirna’ er: gennemsigtigt optimeret design og biofysisk signatur. Nukleinsyrer Res. 35, 433-440.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Leuschner, P. J., Ameres, S. L., Kueng, S. og Martinus, J. (2006). Spaltning af siRNA-passagerstrengen under RISC-samling i humane celler. EMBO Rep. 7, 314-320.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
B. P., Burge, C. B. Og Bartel, D. P. (2005). Konserveret frøparring, ofte flankeret af adenosiner, indikerer, at tusinder af menneskelige gener er mikroRNA-mål. Celle 120, 15-20.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S. og Johnson, J. M. (2005). Microarray-analyse viser, at nogle mikroRNA ‘er nedregulerer et stort antal mål-mRNA’ er. Natur 433, 769-773.
Pubmed Abstrakt / Pubmed Fuld Tekst / CrossRef Fuld Tekst
Lu, J., og Mathias, D. H. (2008). Effektiv siRNA udvælgelse ved hjælp hybridisering termodynamik. Nukleinsyre Res. 36, 640-647.
CrossRef Fuld tekst
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T. og Patel, D. J. (2005). Strukturelt grundlag for 5 ‘ -end-specifik genkendelse af guide-RNA af A. fulgidus PiVi-proteinet. Natur 434, 666-670.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
A., Urlaub, H., Luhrmann, R. Og Tuschl, T. (2002). Enkeltstrengede antisense sirna ‘ er styrer mål RNA-spaltning i RNAi. Celle 110, 563-574.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., P. D. (2005). Passager-streng spaltning Letter samling af siRNA i ago2-holdige RNAi-komplekser. Celle 123, 607-620.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Naito, Y., Yoshimura, J., Morishita, S. og Ui-Tei, K. (2009). siDirect 2.0: opdateret program til at designe funktionelle siRNA med reduceret frø-afhængig off-target effekt. BMC Bioinformatik 10, 392.
CrossRef Fuld tekst
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., vandt, M. Og Kim, Y. J. (2008). AsiDesigner: eksonbaseret siRNA design server overvejer alternativ splejsning. Nukleinsyre Res. 36, V97-V103.
CrossRef Fuld tekst
Rand, T. A., Petersen, S., Du, F. og Vang (2005). Argonaute2 spalter anti-guide-strengen af siRNA under RISC-aktivering. Celle 123, 621-629.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Reynolds, A., Leake, D., Boese, K., Scaringe, S., Marshall, V. S. og Khvorova, A. (2004). Rationelt siRNA-design til RNA-interferens. Nat. Biotechnol. 22, 326–330.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
hutvangner, G., Du, T., S., Aronin, N., P. D. (2003). Asymmetri i samlingen af RNAi-komplekset. Celle 115, 199-208.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Shah, J. K., Garner, H. R., hvid, M. A., Shames, D. S. og Minna, J. D. (2007). sIR: siRNA information resource, et internetbaseret værktøj til siRNA sekvens design og analyse og en open access siRNA database. BMC Bioinformatik 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Fuld tekst
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A. og Saigo, K. (2008). Termodynamisk stabilitet og parring af vand-Crick-basen i frødupleksen er vigtige determinanter for effektiviteten af den sRNA-baserede off-target-effekt. Nukleinsyrer Res. 36, 7100-7109.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamasaki, H., Juni, A., Ueda, R. og Saigo, K. (2004). Retningslinjer for udvælgelse af højeffektive siRNA-sekvenser for pattedyrs-og chick-RNA-interferens. Nukleinsyrer Res. 32, 936-948.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Vert, J.-P., Foveau, N., Lajaunie, C. og Vandenbrouck, Y. (2006). En nøjagtig og fortolkelig model til forudsigelse af siRNA-effektivitet. BMC Bioinformatik 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Fuld tekst
(2004). Siran selection server: en automatiseret siRNA oligonukleotid forudsigelsesserver. Nukleinsyrer Res. 32, 130–134.
Pubmed abstrakt / Pubmed Fuld tekst / CrossRef Fuld tekst
Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Jandina, M., Meister, G., Chen, H. Y., Dauter, S., Tuschl, T. og Patel, D. J. (2005). Krystalstruktur af A. aeolicus argonaute, en stedsspecifik DNA-styret endoribonuklease, giver indsigt i RISC-medieret mRNA-spaltning. Mol. Celle 19, 405-419.
Pubmed Abstrakt / Pubmed Fuld Tekst / CrossRef Fuld Tekst