introduktion
RNA-interferens (RNAi) är en allmänt använd teknik genom vilken små störande RNA (siRNA) nedreglerar en specifik målgen med perfekt komplementär sekvens och lovade att använda i terapeutisk applikation för mänskliga sjukdomar (Castanotto och Rossi, 2009; Ketting, 2011). Även om människan har mer än 20 000 gener är det önskvärt att använda siRNA som är mycket funktionell och inte har några effekter på andra gener än dess specifika mål. I den här artikeln granskar vi en optimerad metod för att designa siRNA baserat på mekanismen för RNAi. Dessutom introducerar vi webbplatser som är öppna för allmänheten för att välja siRNA-sekvenser.
optimerad Design av siRNA
Duplex av 21-nukleotid (nt) RNA med 2 NT 3′ överhäng (siRNA) används vanligtvis för RNAi-experiment. Vid leverans till cellerna införlivas sirna i det RNA-inducerade ljuddämpningskomplexet (RISC) som ett dubbelsträngat RNA. RISC är effektorkomplexet som innehåller Argonaute-protein (sedan) med skivaraktivitet (Hammond et al., 2001; Martinez et al., 2002). SiRNA-styrsträngen som innehåller det termodynamiskt mindre stabila 5 ’ – änden behålls företrädesvis av RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). Passagerarsträngarna i de flesta dubbelsträngade sirna som laddas på RISC klyvs av Ago2-protein och bryts ned (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). De kvarhållna styrsträngparen riktar sig mot mRNA med perfekt komplementär sekvens och förtrycker den genom klyvning av Ago2-protein vid nukleotidposition 10 av siRNA-styrsträngen (Elbashir et al., 2001; Hammond et al., 2001; Martinez et al., 2002). Ett ackumulerat bevis från genomomfattande experiment indikerar emellertid att ett stort antal mRNA med partiella komplementariteter till styrsträngen också reduceras (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Detta fenomen kallas fröberoende off-target-effekt och observeras företrädesvis i mRNA 3’ UTR. Måligenkänningsmekanismen för denna off-target-effekt är känd för att likna den för miRNA-medierad gendämpning (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Transkripten med sekvenser som kompletterar fröområdet placerat 2-8 från 5 ’ – terminalen reduceras huvudsakligen. Fröområdet är känt för att vara beläget på ytan av Ago I en kvasi-spiralform för att fungera som inträdes-eller kärnbildningsplats för små RNA i RISCs (Ma et al., 2005; Yuan et al., 2005). Således identifierar fröregionen först mål-mRNA och bildar därefter perfekt basparning med avsett mål-mRNA och inducerar RNAi av Ago2.
baserat på mekanismen för RNAi anses ett målgenspecifikt siRNA vara valbart enligt följande tre steg.
steg 1: val av funktionell siRNA-sekvens
knockdown-effektiviteten hos siRNAs avslöjas vara mycket beroende av deras sekvenser. Vi förespråkade den empiriskt baserade regeln som föreskrev egenskaperna hos mycket funktionella siRNAs (Ui-Tei et al., 2004) kallas en sådan regel som Ui-Tei-regeln (Figur 1). SiRNA som valts av UI-Tei-regeln uppfyller följande fyra villkor samtidigt: (1) A eller U vid position 1 från 5′ änden av siRNA styrsträng, (2) G eller C vid position 19, (3) AU rikedom (au GHz 4) i positionerna 1-7, och (4) ingen lång GC stretch 10. Med undantag för (4) indikerade vår regel att den funktionella siRNA har asymmetrisk stabilitet i 5′ och 3′ terminaler. Vår experimentella validering med luciferase reporter-analys visade att 98% av siRNA som uppfyller ovanstående villkor minskade uttrycket av luciferase reporter under 33% (Ui-Tei et al., 2004). Andra grupper visade också reglerna för mycket funktionella siRNAs som kallas Reynolds rule (Reynolds et al., 2004) och Amarzguioui rule (Amarzguioui och Prydz, 2004) som sammanfattas i Figur 1. Dessa regler visade också tydligt att funktionella sirna är asymmetriska: en RNA-sträng med instabil 5′ – terminal var effektiv som en styrsträng. Vidare, vanligt i dessa regler, var 5 ’ terminus av funktionell siRNA-styrsträng att föredra att vara A eller U. Det avslöjades senare att resultaten återspeglade de strukturella egenskaperna hos mänsklig Ago2 (Frank et al., 2010). Kristallstrukturen hos en mittdomän från humana AGO2-och NMR-titreringsexperiment visade att nukleotidmonofosfater, AMP och UMP, binder med upp till 30 gånger högre affinitet än antingen CMP eller GMP, vilket ger strukturella bevis för nukleotidspecifika interaktioner i MITTDOMÄNEN för eukaryota ago-proteiner.
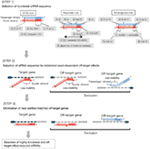
Figur 1. Schematisk representation för att välja funktionella och off-target effektreducerade sirna. Val av mycket funktionell siRNA av UI-Tei, Reynolds, amarzguioui regler, eller kombinationen av dem (Steg 1). Val av siRNA med låg stabilitet i frömålduplexerna (steg 2). Eliminering av siRNAs med nästan perfekt matchade sekvenser till icke-målgener (steg 3). I varje regel anger nukleotidpositionen antalet nukleotider som räknas från 5 ’ – terminalen på styrsträngen.  :
:  A / U vid position 1.
A / U vid position 1.  G / C vid position 19.
G / C vid position 19.  4 till 7 a / Us i positionerna 1-7.
4 till 7 a / Us i positionerna 1-7. 
 :
:  GC-innehåll (30% -52%).
GC-innehåll (30% -52%).  a / u-3 i positionerna 1-5.
a / u-3 i positionerna 1-5.  frånvaro av interna upprepningar.
frånvaro av interna upprepningar.  A vid position 1.
A vid position 1.  A vid position 17.
A vid position 17.  U vid position 10.
U vid position 10.  inte G / C vid position 1.
inte G / C vid position 1. 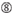 inte G vid position 7.
inte G vid position 7. 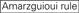 :
:  en asymmetri i stabiliteten hos duplexändarna (mätt som A/U-differentialen för de tre terminalbaspararna i vardera änden av duplexen).
en asymmetri i stabiliteten hos duplexändarna (mätt som A/U-differentialen för de tre terminalbaspararna i vardera änden av duplexen).  G eller C vid position 19.
G eller C vid position 19.  A eller U vid position 1.
A eller U vid position 1.  A eller U vid position 14.
A eller U vid position 14.  inte U vid position 19.
inte U vid position 19.  inte G vid position 1.
inte G vid position 1.
steg 2: Val av siRNA-sekvens med reducerade Off-Target-effekter
för att undvika fröberoende off-target-effekter kan ett tillvägagångssätt vara att välja siRNA-styrsträngen vars frösekvens inte är komplementär till några sekvenser i 3′ UTR av alla icke-riktade gener. Detta tillvägagångssätt har dock visat sig vara omöjligt, eftersom mänskliga sirna med den mest sällsynta sju-nt-frösekvensen fortfarande hade frökomplementariteter med flera icke-riktade mRNA. Så vi har letat efter de regler som styr siRNAs förmåga att inducera fröberoende off-target-effekt och avslöjade att effektiviteten hos off-target-effekten är starkt korrelerad med den termodynamiska stabiliteten hos duplexen som bildas mellan fröregionen av siRNA guide strand och dess mål mRNA (Ui-Tei et al., 2008). Smälttemperaturen (Tm), en av de termodynamiska parametrarna för bildandet av RNA-duplex, visade stark positiv korrelation med induktionen av fröberoende off-target-effekter. Således bör val av siRNA med låg Tm av frömålduplexen minimera fröberoende off-target-tystnad (Figur 1). Tm på 21,5 C kan fungera som riktmärke, vilket diskriminerar de nästan off-target-fria frösekvenserna från de off-target-positiva. Dessutom, eftersom off-target-effekten kan orsakas av inte bara av styrsträngen utan också av passagerarsträngen, är sirna vars frömål Tm är tillräckligt lågt för båda strängarna gynnsamma.
steg 3: Eliminering av nästan perfekt matchade off-Target-gener
även när TM-värdet för frömålduplexen är tillräckligt lågt kan målgenens tystnad fortfarande äga rum om icke-fröområdet är helt komplementärt. Därför eliminerades i det tredje steget siRNAs som har nästan perfekta matchningar till andra icke-riktade transkript (Figur 1).
Sirna Design Software
vi presenterade siRNA Design software, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figur 2), som tillhandahåller funktionell, målspecifik siRNA-designprogramvara enligt ovan nämnda förfaranden (Naito et al., 2009). I standardparametern kan siRNAs som uppfyller Ui-Tei-regeln väljas. När kandidatfunktionella sirna kan bilda frömålduplexer med TM-värden under 21.5 C, och deras 19-nt-regioner som spänner över positionerna 2-20 i båda strängarna har minst två felmatchningar till andra icke-riktade transkript, kan siDirect 2.0 designa minst en kvalificerad siRNA för >94% av humana mRNA-sekvenser i RefSeq.

Figur 2. Skärm utsikt över siDirect 2.0 siRNA design programvara. (A) översta sidan (http://siDirect2.RNAi.jp/). (B) valfria parametrar för siRNA design. (C) resultatsida. (D) detaljerad lista över kandidater utanför målet med nästan perfekta matcher. Inriktningen mellan varje off-target-transkript och siRNA-sekvensen visualiserar positionerna för felaktigheter.
annan programvara för att välja funktionella sirna var öppen för allmänheten som visas i Tabell 1. I många av dem, Ui-Tei regel (Ui-Tei et al., 2004), Reynolds regel (Reynolds et al., 2004), Amarzguioui-regeln (Amarzguioui och Prydz, 2004), Tuschl-regeln (Elbashir et al., 2002), och kombinationen av dem användes ofta och i stor utsträckning. För att eliminera de nästan perfekta matchade icke-målgenerna användes BLAST search för homologisökning i flera program. Men eftersom BLAST-sökning inte är så exakt för korta sekvenser som siRNAs, siDirect, WU-BLAST och Bowtie, som är mycket exakta homologisökmotor för korta sekvenser används ofta. Bland dem kan siDirect 2.0 ge de mest exakta resultaten. Dessutom överväger en del av programvaran ytterligare funktioner, såsom mRNA sekundär struktur (Ladunga, 2007; Lu och Mathews, 2008), alternativ skarvning (Park et al., 2008), eller motivsekvensen genom vilken immunsvaret av ett RNA-virus kan uppstå (Gong et al., 2008). Dessa funktioner beaktas inte i siDirect 2.0. Så, för att ta hänsyn till dessa funktioner, siRNAs vanligen väljs av siDirect 2.0 och andra lämpliga program kan ge optimalt resultat.
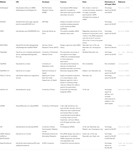
Tabell 1. Små interfererande RNA-designprogram.
dessutom är det praktiskt taget viktigt att överväga att använda två eller flera sirna som riktar sig mot olika platser i en avsedd målgen, eftersom knockdown-effekterna av en avsedd målgen antas vara vanliga men off-target-effekterna kommer sannolikt att vara olika mellan sirna.
slutsats
effekten av varje siRNA är känd för att vara mycket varierar beroende på dess sekvens i däggdjursceller, och endast en begränsad bråkdel av slumpmässigt utformade sirna är funktionell. Dessutom uppstår off-target-ljuddämpande effekter när siRNA har partiell komplementaritet i fröområdet med oavsiktliga gener. Här, baserat på RNAi-maskinerna, beskrev vi den rationella utformningen av funktionella, off-target-effektreducerade sirna, som förväntas slå ner en målgen-specifikt.
intressekonflikt uttalande
författarna förklarar att forskningen genomfördes i avsaknad av kommersiella eller finansiella relationer som kan tolkas som en potentiell intressekonflikt.
bekräftelser
detta arbete stöddes delvis av bidrag från Ministeriet för utbildning, kultur, sport, vetenskap och teknik i Japan (MEXT) och Cell Innovation Project (MEXT) och Core Research Project for Private University; matchande fondbidrag till Kumiko Ui-Tei.
Amarzguioui, M. och Prydz, H. (2004). En algoritm för val av funktionella siRNA-sekvenser. Biochem. Biophys. Res. Commun. 316, 1050–1058.
PubMed Abstrakt / Pubmed Fulltext / CrossRef Fulltext
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S. och Khvorova, A. (2006). 3 ’ UTR frömatchningar, men inte övergripande identitet, är associerade med RNAi off-mål. Nat. Metoder 3, 199-204.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Castanotto, D. och Rossi, J. J. (2009). Löften och fallgropar av RNA-interferensbaserade terapier. Natur 457, 426-433.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
krita, AM och Sonnhammer, ell (2008). siRNA specificitet söka införliva mismatch toleransdata. Bioinformatik 24, 1316-1317.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Cui, W., Ning, J., Naik, U. P. och Duncan, M. K. (2004). OptiRNAi och RNAi designverktyg. Dator. Metoder Program Biomed. 75, 67–73.
PubMed Abstrakt / Pubmed Fulltext / CrossRef Fulltext
Ding, Y., Chan, C. Y., och Lawrence, C. E. (2004). Sfold webbserver för statistisk vikning och rationell design av nukleinsyror. Nukleinsyror Res. 32, W135-W141.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K. och Tuschl, T. (2001). Duplex av 21-nukleotid rna förmedlar RNA-interferens i odlade däggdjursceller. Natur 411, 494-498.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Elbashir, W. M., Harborth, J., Weber, K. och Tuschl, T. (2002). Analys av genfunktion i somatiska däggdjursceller med hjälp av små störande RNA. Metoder 26, 199-213.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Frank, F., Sonenberg, N. och Nagar, B. (2010). Strukturell grund för 5 ’ – nukleotidbasspecifikt erkännande av guide RNA av humant AGO2. Natur 465, 818-822.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S. och Li, Tongbin. (2008). siDRM: och effektivt och allmänt tillämpligt online siRNA designverktyg. Bioinformatik 24, 2405-2406.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P. och Bartel, D. P. (2007). MikroRNA riktar sig mot specificitet hos däggdjur: determinanter utöver fröparning. Mol. Cell 27, 91-105.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R. och Hannon, G. J. (2001). Argonaute 2 länk mellan genetiska och biokemiska analyser av RNAi. Vetenskap 293, 1146-1150.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Henschel, A., Buchholz, F. och Habermann, B. (2004). DEQOR: ett webbaserat verktyg för design och kvalitetskontroll av siRNAs. Nukleinsyror Res. 32, W113-W120.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Horn, T., Sandmann, T. och Boutros, M. (2010). Design och utvärdering av genomomfattande bibliotek för RNA-interferensskärmar. Genome Biol. 11, R61.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Jackson, A. L., Bartz, Sr, Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G. och Linsley, P. S. (2003). Uttrycksprofilering avslöjar off-target genreglering av RNAi. Nat. Bioteknol. 21, 635–637.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L. och Linsley, P. S. (2006). Utbredd siRNA” off-target ” transkript tystnad medierad av fröregionsekvenskomplementaritet. RNA 12, 1179-1187.
PubMed Abstrakt / Pubmed Fulltext / CrossRef Fulltext
Ketting, R. F. (2011). De många ansikten RNAi. Dev. Cell 15, 148-161.
CrossRef Fullständig Text
Khvorova, A., Reynolds, A. och Jayasena, S. D. (2003). Funktionella siRNAs och miRNAs uppvisar strängförspänning. Cell 115, 209-216.
PubMed Abstrakt / Pubmed Fulltext / CrossRef Fulltext
Ladunga, I. (2007). Mer komplett gentystning av färre sirna: transparent optimerad design och biofysisk signatur. Nukleinsyror Res. 35, 433-440.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Leuschner, PJ, Ameres, sl, Kueng, S. och Martinez, J. (2006). Klyvning av siRNA-passagerarsträngen under RISC-montering i mänskliga celler. EMBO rep. 7, 314-320.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Lewis, B. P., Burge, C. B. Och Bartel, D. P. (2005). Konserverad fröparning, ofta flankerad av adenosiner, indikerar att tusentals mänskliga gener är mikroRNA-mål. Cell 120, 15-20.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Lim, lp, Lau, NC, Garrett-Engele, P., Grimson, A., Schelter, JM, Castle, J., Bartel, DP, Linsley, Psoch Johnson, JM (2005). Mikroarrayanalys visar att vissa mikroRNA nedreglerar ett stort antal mål-mRNA. Natur 433, 769-773.
PubMed Abstrakt / Pubmed Fulltext / CrossRef Fulltext
Lu, Z. J., och Mathews, D. H. (2008). Effektiv siRNA val med hjälp av hybridisering termodynamik. Nukleinsyra Res. 36, 640-647.
CrossRef Fullständig Text
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T. och Patel, DJ (2005). Strukturell grund för 5 ’ -end-specifikt erkännande av guide RNA av A. fulgidus piwi-proteinet. Natur 434, 666-670.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R. och Tuschl, T. (2002). Enkelsträngad antisense siRNAs styr mål-RNA-klyvning i RNAi. Cell 110, 563-574.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P. och Zamore, P. D. (2005). Klyvning av passagerarsträngar underlättar montering av siRNA i Ago2-innehållande RNAi-enzymkomplex. Cell 123, 607-620.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Naito, Y., Yoshimura, J., Morishita, S. och Ui-Tei, K. (2009). siDirect 2.0: uppdaterad programvara för att designa funktionell siRNA med reducerad fröberoende off-target-effekt. BMC bioinformatik 10, 392.
CrossRef Fullständig Text
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M. och Kim, Y. J. (2008). AsiDesigner: exon-baserade siRNA design server överväger alternativ skarvning. Nukleinsyra Res. 36, W97-W103.
CrossRef Fullständig Text
Rand, T. A., Petersen, S., Du, F. och Wang, X. (2005). Argonaute2 klyver anti-styrsträngen av siRNA under RISC-aktivering. Cell 123, 621-629.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S. och Khvorova, A. (2004). Rationell siRNA-design för RNA-interferens. Nat. Bioteknol. 22, 326–330.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N. och Zamore, P. D. (2003). Asymmetri vid montering av RNAi-enzymkomplexet. Cell 115, 199-208.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Shah, JK, Garner, HR, White, ma, Shames, DS och Minna, jd (2007). sIR: siRNA information resource, ett webbaserat verktyg för siRNA sekvensdesign och analys och en open access siRNA-databas. BMC bioinformatik 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Full Text
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A. och Saigo, K. (2008). Termodynamisk stabilitet och Watoson-Crick-basparning i fröduplexen är viktiga determinanter för effektiviteten hos den sRNA-baserade off-target-effekten. Nukleinsyror Res. 36, 7100-7109.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. och Saigo, K. (2004). Riktlinjer för val av mycket effektiva siRNA sekvenser för däggdjur och chick RNA-interferens. Nukleinsyror Res. 32, 936-948.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Vert, J.-P., Foveau, N., Lajaunie, C. och Vandenbrouck, Y. (2006). En exakt och tolkbar modell för siRNA efficacy prediction. BMC bioinformatik 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Full Text
Yuan, B., Latek, R., Rossbach, M., Tuschl, T. och Lewitter, F. (2004). siRAN selection server: en automatiserad siRNA oligonukleotid förutsägelse server. Nukleinsyror Res. 32, W130-W134.
PubMed Abstrakt / Pubmed fulltext / CrossRef fulltext
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T. och Patel, DJ (2005). Kristallstruktur av A. aeolicus argonaute, ett platsspecifikt DNA-styrt endoribonukleas, ger insikter i RISC-medierad mRNA-klyvning. Mol. Cell 19, 405-419.
PubMed Abstrakt / Pubmed Fulltext / CrossRef Fulltext