- Introduction
- Optimiertes Design von siRNA
- SCHRITT 1: Auswahl der funktionellen siRNA-Sequenz
- SCHRITT 2: Auswahl der siRNA-Sequenz mit reduzierten Off-Target-Effekten
- SCHRITT 3: Elimination von nahezu perfekt aufeinander abgestimmten Off-Target-Genen
- siRNA Design Software
- Schlussfolgerung
- Interessenkonflikterklärung
- Danksagungen
Introduction
Die RNA-Interferenz (RNAi) ist eine weit verbreitete Technik, mit der kleine interferierende RNA (siRNA) ein spezifisches Zielgen mit perfekter komplementärer Sequenz herunterreguliert und in der therapeutischen Anwendung für menschliche Krankheiten eingesetzt werden kann (Castanotto und Rossi, 2009; Ketting, 2011). Obwohl der Mensch mehr als 20.000 Gene hat, ist es wünschenswert, siRNA zu verwenden, die hochfunktionell ist und keine Auswirkungen auf andere Gene als ihr spezifisches Ziel hat. In diesem Artikel, Wir überprüfen eine optimierte Methode zum Entwerfen von siRNA basierend auf dem Mechanismus von RNAi. Darüber hinaus stellen wir die öffentlich zugänglichen Websites zur Auswahl von siRNA-Sequenzen vor.
Optimiertes Design von siRNA
Duplexe von 21-Nukleotid (nt) -RNA mit 2 nt 3′-Überhängen (siRNA) werden normalerweise für RNAi-Experimente verwendet. Bei der Abgabe in die Zellen werden siRNAs als doppelsträngige RNA in den RNA-induzierten Silencing-Komplex (RISC) eingebaut. RISC ist der Effektorkomplex, der Argonaute-Protein (Ago) mit hoher Aktivität enthält (Hammond et al., 2001; Martinez et al., 2002). Der siRNA-Führungsstrang mit dem thermodynamisch weniger stabilen 5′-Ende wird bevorzugt von RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). Die Passagierstränge der meisten auf RISC geladenen doppelsträngigen siRNAs werden durch Ago2-Protein gespalten und abgebaut (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). Der zurückgehaltene Führungsstrang paart Ziel-mRNA mit perfekt komplementärer Sequenz und unterdrückt sie durch Spaltung durch Ago2-Protein an Nukleotidposition 10 des siRNA-Führungsstrangs (Elbashir et al., 2001; Hammond et al., 2001; Martinez et al., 2002). Eine akkumulierte Evidenz aus genomweiten Experimenten zeigt jedoch, dass eine große Anzahl von mRNAs mit partieller Komplementarität zum Führungsstrang ebenfalls reduziert ist (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Dieses Phänomen wird als Seed-abhängiger Off-Target-Effekt bezeichnet und vorzugsweise in mRNA 3′-UTRs beobachtet. Es ist bekannt, dass der Zielerkennungsmechanismus dieses Off-Target-Effekts dem des miRNA-vermittelten Gen-Silencings ähnelt (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Die Transkripte mit Sequenzen, die komplementär zur Seed-Region 2-8 vom 5′-Terminal positioniert sind, werden hauptsächlich reduziert. Es ist bekannt, dass sich die Seed-Region auf der Oberfläche von Ago in einer quasi-helikalen Form befindet, um als Eintritts- oder Nukleationsstelle für kleine RNAs in den RISC zu dienen (Ma et al., 2005; Yuan et al., 2005). Somit identifiziert die Seed-Region zuerst die Ziel-mRNAs und bildet anschließend eine perfekte Basenpaarung mit der Ziel-mRNA und induziert RNAi durch Ago2.
Basierend auf dem Mechanismus von RNAi wird eine zielgenspezifische siRNA gemäß den folgenden drei Schritten als auswählbar angesehen.
SCHRITT 1: Auswahl der funktionellen siRNA-Sequenz
Es zeigt sich, dass die Knockdown-Effizienz von siRNAs stark von ihren Sequenzen abhängt. Wir befürworteten die empirisch fundierte Regel, die die Eigenschaften hochfunktioneller siRNAs vorschreibt (Ui-Tei et al., 2004) wird eine solche Regel als Ui-Tei-Regel bezeichnet (Abbildung 1). Die von Ui-Tei ausgewählte siRNA erfüllt gleichzeitig die folgenden vier Bedingungen: (1) A oder U an Position 1 vom 5′-Terminus des siRNA-Führungsstrangs, (2) G oder C an Position 19, (3) AU-Reichtum (AU ≥4) an den Positionen 1-7 und (4) keine lange GC-Dehnung ≥10. Mit Ausnahme von (4) zeigte unsere Regel, dass die funktionelle siRNA eine asymmetrische Stabilität in 5′- und 3′-Terminals aufweist. Unsere experimentelle Validierung mit dem Luciferase Reporter Assay zeigte, dass 98% der siRNAs, die die oben genannten Bedingungen erfüllten, die Expression von Luciferase Reporter unter 33% reduzierten (Ui-Tei et al., 2004). Andere Gruppen zeigten auch die Regeln hochfunktioneller siRNAs, die als Reynolds-Regel bezeichnet werden (Reynolds et al., 2004) und Amarzguioui-Regel (Amarzguioui und Prydz, 2004), wie in Abbildung 1 zusammengefasst. Diese Regeln zeigten auch deutlich, dass funktionelle siRNAs asymmetrisch sind: Ein RNA-Strang mit instabilem 5′-Terminal war als Führungsstrang wirksam. Darüber hinaus war der in diesen Regeln übliche 5′-Terminus des funktionellen siRNA-Führungsstrangs bevorzugt A oder U. Es zeigte sich später, dass die Ergebnisse die strukturellen Merkmale von humanem Ago2 widerspiegelten (Frank et al., 2010). Die Kristallstruktur einer mittleren (mittleren) Domäne aus humanen AGO2- und NMR-Titrationsexperimenten zeigte, dass Nukleotidmonophosphate, AMP und UMP, mit einer bis zu 30-fach höheren Affinität als CMP oder GMP binden, was strukturelle Beweise für nukleotidspezifische Wechselwirkungen in der mittleren Domäne von eukaryotischen AGO-Proteinen liefert.
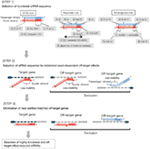
Abbildung 1. Schematische Darstellung zur Selektion funktioneller und Off-Target effektreduzierter siRNAs. Selektion hochfunktioneller siRNA nach Ui-Tei-, Reynolds-, Amarzguioui-Regeln oder deren Kombination (SCHRITT 1). Selektion von siRNAs mit geringer Stabilität in den Seed-Target-Duplexen (SCHRITT 2). Eliminierung von siRNAs mit nahezu perfekt aufeinander abgestimmten Sequenzen zu Nichtzielgenen (SCHRITT 3). In jeder Regel gibt die Nukleotidposition die Anzahl der Nukleotide an, die vom 5′-Terminal des Führungsstrangs gezählt werden.  :
:  A/U an Position 1.
A/U an Position 1.  G/C an Position 19.
G/C an Position 19.  4 bis 7 A/Us in den Positionen 1-7.
4 bis 7 A/Us in den Positionen 1-7. 
 :
:  GC-Gehalt (30%-52%).
GC-Gehalt (30%-52%).  A/U ≥ 3 an den Positionen 1-5.
A/U ≥ 3 an den Positionen 1-5.  Fehlen interner Wiederholungen.
Fehlen interner Wiederholungen.  A an Position 1.
A an Position 1.  A an Position 17.
A an Position 17.  U an Position 10.
U an Position 10.  Nicht G/C an Position 1.
Nicht G/C an Position 1. 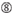 Nicht G an Position 7.
Nicht G an Position 7. 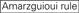 :
:  Eine Asymmetrie in der Stabilität der Duplexenden (gemessen als A / U-Differenz der drei Anschlussbasispaare an beiden Enden des Duplexes).
Eine Asymmetrie in der Stabilität der Duplexenden (gemessen als A / U-Differenz der drei Anschlussbasispaare an beiden Enden des Duplexes).  G oder C an Position 19.
G oder C an Position 19.  A oder U an Position 1.
A oder U an Position 1.  A oder U an Position 14.
A oder U an Position 14.  Nicht U an Position 19.
Nicht U an Position 19.  Nicht G an Position 1.
Nicht G an Position 1.
SCHRITT 2: Auswahl der siRNA-Sequenz mit reduzierten Off-Target-Effekten
Um Seed-abhängige Off-Target-Effekte zu vermeiden, kann ein Ansatz darin bestehen, den siRNA-Führungsstrang auszuwählen, dessen Seed-Sequenz nicht komplementär zu irgendwelchen Sequenzen in der 3’UTR aller nicht zielgerichteten Gene ist. Dieser Ansatz hat sich jedoch als unmöglich erwiesen, da menschliche siRNAs mit der seltensten Seven-nt-Seedsequenz immer noch Seed-Komplementaritäten mit mehreren nicht zielgerichteten mRNAs aufwiesen. Daher haben wir nach den Regeln gesucht, die die Fähigkeit von siRNAs regeln, einen Seed-abhängigen Off-Target-Effekt zu induzieren, und gezeigt, dass die Effizienz des Off-Target-Effekts in hohem Maße mit der thermodynamischen Stabilität des Duplexes korreliert, der zwischen der Seed-Region des siRNA-Führungsstrangs und seiner Ziel-mRNA gebildet wird (Ui-Tei et al., 2008). Die Schmelztemperatur (Tm), einer der thermodynamischen Parameter für die Bildung von RNA-Duplex, zeigte eine starke positive Korrelation mit der Induktion von Seed-abhängigen Off-Target-Effekten. Daher sollte die Auswahl der siRNAs mit geringer Tm des Seed-Target-Duplexes die Seed-abhängige Off-Target-Stummschaltung minimieren (Abbildung 1). Der Tm von 21,5 °C kann als Benchmark dienen, der die fast off-Target-freien Seed-Sequenzen von den Off-Target-positiven unterscheidet. Da ferner der Off-Target-Effekt nicht nur durch den Führungsstrang, sondern auch durch den Passagierstrang hervorgerufen werden kann, sind siRNAs günstig, deren Seed-Target-Tm für beide Stränge ausreichend niedrig ist.
SCHRITT 3: Elimination von nahezu perfekt aufeinander abgestimmten Off-Target-Genen
Selbst wenn der Tm-Wert des Seed-Target-Duplexes ausreichend niedrig ist, kann die Zielgen-Stummschaltung noch stattfinden, wenn die Nicht-Seed-Region vollständig komplementär ist. Daher wurden im dritten Schritt siRNAs eliminiert, die nahezu perfekte Übereinstimmungen mit anderen nicht zielgerichteten Transkripten aufweisen (Abbildung 1).
siRNA Design Software
Wir präsentierten siRNA Design Software, siDirect 2.0 (http://siDirect2.RNAi.jp/; Abbildung 2), die eine funktionale, zielspezifische siRNA-Designsoftware gemäß den oben genannten Verfahren bereitstellt (Naito et al., 2009). Im Default-Parameter können siRNAs ausgewählt werden, die die Ui-Tei-Regel erfüllen. Wenn die kandidatenfunktionalen siRNAs Seed-Target-Duplexe mit Tm-Werten unter 21,5 ° C bilden könnten und ihre 19-nt-Regionen, die die Positionen 2-20 beider Stränge überspannen, mindestens zwei Fehlpaarungen zu anderen nicht zielgerichteten Transkripten aufweisen, kann siDirect 2.0 mindestens eine qualifizierte siRNA für > 94% der humanen mRNA-Sequenzen in RefSeq entwerfen.

Abbildung 2. Bildschirmansichten von siDirect 2.0 siRNA Design Software. (A) Startseite (http://siDirect2.RNAi.jp/). (B) Optionale Parameter für das siRNA-Design. (C) Ergebnisseite. (D) Detaillierte Liste der nicht zielgerichteten Kandidaten mit nahezu perfekten Übereinstimmungen. Die Ausrichtung zwischen jedem Off-Target-Transkript und der siRNA-Sequenz visualisiert die Positionen von Fehlanpassungen.
Andere Software zur Auswahl funktioneller siRNAs war öffentlich zugänglich, wie in Tabelle 1 gezeigt. In vielen von ihnen gilt die Ui-Tei-Regel (Ui-Tei et al., 2004), Reynolds-Regel (Reynolds et al., 2004), Amarzguioui-Regel (Amarzguioui und Prydz, 2004), Tuschl-Regel (Elbashir et al., 2002), und die Kombination von ihnen wurden häufig und weit verbreitet. Um die nahezu perfekt aufeinander abgestimmten Nichtzielgene zu eliminieren, wurde BLAST Search für die Homologiesuche in mehreren Programmen verwendet. Da jedoch die BLAST-Suche für kurze Sequenzen wie siRNAs, siDirect, WU-BLAST und Bowtie nicht so genau ist, werden häufig hochgenaue Homologie-Suchmaschinen für kurze Sequenzen verwendet. Unter ihnen kann siDirect 2.0 die genauesten Ergebnisse bringen. Darüber hinaus berücksichtigen einige der Software die zusätzlichen Funktionen, wie mRNA-Sekundärstruktur (Ladunga, 2007; Lu und Mathews, 2008), alternatives Spleißen (Park et al., 2008), oder die Motivsequenz, mit der die Immunantwort durch ein RNA-Virus auftreten könnte (Gong et al., 2008). Diese Funktionen werden in siDirect 2.0 nicht berücksichtigt. Um diese Merkmale zu berücksichtigen, können siRNAs, die üblicherweise von siDirect 2.0 und den anderen geeigneten Softwareprogrammen ausgewählt werden, ein optimales Ergebnis liefern.
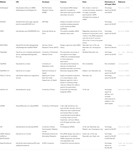
Tabelle 1. Kleine störende RNA-Design-Software-Programme.
Darüber hinaus ist es eine wichtige Überlegung, zwei oder mehr siRNAs zu verwenden, die auf verschiedene Stellen in einem beabsichtigten Zielgen abzielen, da die Knockdown-Effekte eines beabsichtigten Zielgens häufig sein sollen, aber die Off-Target-Effekte sind wahrscheinlich zwischen siRNAs unterschiedlich.
Schlussfolgerung
Es ist bekannt, dass die Wirksamkeit jeder siRNA in Abhängigkeit von ihrer Sequenz in Säugetierzellen stark variiert, und nur ein begrenzter Anteil zufällig konstruierter siRNAs ist funktionsfähig. Darüber hinaus treten Off-Target-Silencing-Effekte auf, wenn die siRNA eine partielle Komplementarität in der Samenregion mit unbeabsichtigten Genen aufweist. Hier, basierend auf der RNAi-Maschinerie, Wir haben das rationale Design funktioneller beschrieben, Off-Target-effektreduzierte siRNAs, von denen erwartet wird, dass sie ein Zielgen spezifisch niederschlagen.
Interessenkonflikterklärung
Die Autoren erklären, dass die Forschung in Abwesenheit von kommerziellen oder finanziellen Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Danksagungen
Diese Arbeit wurde teilweise durch Zuschüsse des japanischen Ministeriums für Bildung, Kultur, Sport, Wissenschaft und Technologie (MEXT) und des Cell Innovation Project (MEXT) sowie des Core Research Project für private Universitäten unterstützt.
Amarzguioui, M. und Prydz, H. (2004). Ein Algorithmus zur Auswahl funktioneller siRNA-Sequenzen. Biochem. In: Biophys. Res. Commun. 316, 1050–1058.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S. und Khvorova, A. (2006). 3′ UTR-Seed-Matches, aber keine Gesamtidentität, sind mit RNAi-Off-Targets verbunden. Nat. Methoden 3, 199-204.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Castanotto, D. und Rossi, JJ (2009). Die Versprechungen und Fallstricke von RNA-Interferenz-basierten Therapeutika. Natur 457, 426-433.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Kreide, A. M. und Sonnhammer, ELL (2008). siRNA-Spezifitätssuche mit Mismatch-Toleranzdaten. Bioinformatik 24, 1316-1317.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Cui, W., Ning, J., Naik, U. P. und Duncan, M. K. (2004). OptiRNAi und RNAi Design Tool. In: Comput. Methoden Programme Biomed. 75, 67–73.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Ding, Y., Chan, C. Y., und Lawrence, CE (2004). Sfold Webserver für statistische Faltung und rationales Design von Nukleinsäuren. Nukleinsäuren Res. 32, W135-W141.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Elbashir, SM, Harborth, J., Lendeckel, W., Yalcin, A., Weber, K. und Tuschl, T. (2001). Duplexe von 21-Nukleotid-RNAs vermitteln RNA-Interferenz in kultivierten Säugetierzellen. Natur 411, 494-498.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Elbashir, W. M., Harborth, J., Weber, K. und Tuschl, T. (2002). Analyse der Genfunktion in somatischen Säugetierzellen mit kleinen interferierenden RNAs. Methoden 26, 199-213.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Frank, F., Sonenberg, N. und Nagar, B. (2010). Strukturelle Basis für die 5′-nukleotidbasenspezifische Erkennung von Guide-RNA durch humanes AGO2. Natur 465, 818-822.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
In der Praxis ist die Verwendung von Cookies nicht gestattet. (2008). siDRM: und effektives und allgemein anwendbares Online-siRNA-Design-Tool. Bioinformatik 24, 2405-2406.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P. und Bartel, D. P. (2007). microRNA-Targeting-Spezifität bei Säugetieren: Determinanten jenseits der Samenpaarung. Mol. Zelle 27, 91-105.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Hammond, SM, Boettcher, S., Caudy, AA, Kobayashi, R. und Hannon, GJ (2001). Argonaute 2 Verbindung zwischen genetischen und biochemischen Analysen von RNAi. Wissenschaft 293, 1146-1150.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Henschel, A., Buchholz, F., und Habermann, B. (2004). DEQOR: ein webbasiertes Tool für das Design und die Qualitätskontrolle von siRNAs. Nukleinsäuren Res. 32, W113-W120.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Horn, T., Sandmann, T. und Boutros, M. (2010). Design und Evaluation von genomweiten Bibliotheken für RNA-Interferenz-Screens. In: Genome Biol. 11, R61.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Jackson, A. L., Bartz, SR., Shelter, J., Kobayashi, SV, Burchard, J., Mao, M., Li, B., Cavet, G. und Linsley, PS (2003). Expressionsprofil zeigt Off-Target-Genregulation durch RNAi. Nat. In: Biotechnol. 21, 635–637.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L. und Linsley, PS (2006). Weit verbreitete siRNA- „Off-Target“ -Transkript-Stummschaltung, vermittelt durch Komplementarität der Seed-Region-Sequenz. RNA 12, 1179-1187.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Ketting, R. F. (2011). Die vielen Gesichter von RNAi. Dev. Zelle 15, 148-161.
Querverweis Volltext
Khvorova, A., Reynolds, A. und Jayasena, SD (2003). Funktionelle siRNAs und miRNAs weisen eine Strangverzerrung auf. Zelle 115, 209-216.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Ladunga, I. (2007). Vollständigeres Gen-Silencing durch weniger siRNAs: transparentes optimiertes Design und biophysikalische Signatur. Nukleinsäuren Res. 35, 433-440.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Leuschner, PJ, Ameres, SL, Kueng, S. und Martinez, J. (2006). Spaltung des siRNA-Passagierstrangs während der RISC-Assemblierung in menschlichen Zellen. EMBO Rep. 7, 314-320.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Lewis, B. P., Burge, C. B. und Bartel, D. P. (2005). Konservierte Samenpaarung, oft flankiert von Adenosinenzeigt an, dass Tausende menschlicher Gene microRNA-Ziele sind. Zelle 120, 15-20.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S. und Johnson, J. M. (2005). Die Microarray-Analyse zeigt, dass einige microRNAs eine große Anzahl von Ziel-mRNAs herunterregulieren. Natur 433, 769-773.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Lu, Z. J., und Mathews, DH (2008). Effiziente siRNA-Selektion mittels Hybridisierungsthermodynamik. Nucleic Acid Res. 36, 640-647.
Querverweis Volltext
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T. und Patel, DJ (2005). Strukturelle Basis für die 5′-endspezifische Erkennung von Guide-RNA durch das A. fulgidus piwi Protein. Natur 434, 666-670.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R., und Tuschl, T. (2002). Einzelsträngige Antisense-siRNAs steuern die Ziel-RNA-Spaltung in RNAi. Zelle 110, 563-574.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P. und Zamore, P. D. (2005). Die Mehrstrangspaltung erleichtert den Zusammenbau von siRNA in Ago2-haltige RNAi-Enzymkomplexe. Zelle 123, 607-620.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Naito, Y., Yoshimura, J., Morishita, S. und Ui-Tei, K. (2009). siDirect 2.0: aktualisierte Software zum Entwerfen funktioneller siRNA mit reduziertem Seed-abhängigem Off-Target-Effekt. BMC Bioinformatik 10, 392.
Querverweis Volltext
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Gewonnen, M., und Kim, YJ (2008). AsiDesigner: Exon-basierter siRNA-Designserver, der alternatives Spleißen in Betracht zieht. Nukleinsäure Res. 36, W97-W103.
Querverweis Volltext
Rand, T. A., Petersen, S., Du, F. und Wang, X. (2005). Argonaute2 spaltet den Anti-Guide-Strang der siRNA während der RISC-Aktivierung. Zelle 123, 621-629.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S. und Khvorova, A. (2004). Rationales siRNA-Design für RNA-Interferenz. Nat. In: Biotechnol. 22, 326–330.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Schwarz, DS, Hutvangner, G., Du, T., Xu, Z., Aronin, N. und Zamore, PD (2003). Asymmetrie beim Aufbau des RNAi-Enzymkomplexes. Zelle 115, 199-208.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Shah, JK, Garner, HR, White, MA, Shames, DS und Minna, JD (2007). sIR: siRNA Information Resource, ein webbasiertes Tool für das Design und die Analyse von siRNA-Sequenzen und eine Open-Access-siRNA-Datenbank. BMC Bioinformatik 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Volltext
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A. und Saigo, K. (2008). Thermodynamische Stabilität und Watoson-Crick-Basenpaarung im Seed-Duplex sind wichtige Determinanten für die Effizienz des sRNA-basierten Off-Target-Effekts. Nukleinsäuren Res. 36, 7100-7109.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. und Saigo, K. (2004). Richtlinien für die Auswahl hochwirksamer siRNA-Sequenzen für die RNA-Interferenz von Säugetieren und Küken. Nukleinsäuren Res. 32, 936-948.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Vert, J.-P., Foveau, N., Lajaunie, C. und Vandenbrouck, Y. (2006). Ein genaues und interpretierbares Modell für die Vorhersage der siRNA-Wirksamkeit. BMC Bioinformatik 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Volltext
Yuan, B., Latek, R., Rossbach, M., Tuschl, T. und Lewitter, F. (2004). siRAN Selection Server: ein automatisierter siRNA-Oligonukleotid-Vorhersage-Server. Nukleinsäuren Res. 32, W130-W134.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T. und Patel, DJ (2005). Die Kristallstruktur von A. aeolicus argonaute, einer ortsspezifischen DNA-geführten Endoribonuklease, liefert Einblicke in die RISC-vermittelte mRNA-Spaltung. Mol. Zelle 19, 405-419.
Pubmed Zusammenfassung / Pubmed Volltext / CrossRef Volltext