- Introducere
- design optimizat al siARN
- pasul 1: Selectarea secvenței funcționale de siARN
- pasul 2: Selectarea secvenței siARN cu efecte Off-țintă reduse
- Pasul 3: Eliminarea genelor Off-țintă aproape perfecte potrivite
- software de proiectare siARN
- concluzie
- Declarație privind conflictul de interese
- mulțumiri
Introducere
interferența ARN (RNAi) este o tehnică larg utilizată prin care ARN-ul interferent mic (siRNA) reglează în jos o genă țintă specifică cu secvență complementară perfectă și a promis că va fi utilizată în aplicații terapeutice pentru bolile umane (Castanotto și Rossi, 2009; Ketting, 2011). Deși omul are mai mult de 20.000 de gene, este de dorit să se utilizeze siARN care este extrem de funcțional și nu are efecte asupra altor gene decât ținta sa specifică. În acest articol, analizăm o metodă optimizată de proiectare a siARN bazată pe mecanismul RNAi. În plus, prezentăm Site-urile web deschise publicului pentru selectarea secvențelor siARN.
design optimizat al siARN
Duplexuri de ARN 21-nucleotide (nt) cu 2 NT 3′ consolele (siARN) este de obicei folosit pentru experimentele RNAi. La livrarea în celule, siRNAs sunt încorporate în complexul de tăcere indus de ARN (RISC) ca ARN dublu catenar. RISC este complexul efector care conține proteine Argonaute (Ago) cu activitate de feliere (Hammond și colab., 2001; Martinez și colab., 2002). Firul de ghidare siRNA care conține capătul 5’mai puțin stabil din punct de vedere termodinamic este păstrat preferențial de RISC (Khvorova și colab., 2003; Schwarz și colab., 2003; Ui-Tei și colab., 2004). Firele de pasageri ale majorității sirn-urilor dublu catenare încărcate pe RISC sunt scindate de proteina Ago2 și degradate (Matranga și colab., 2005; Rand și colab., 2005; Leuschner și colab., 2006). Perechile de catene de ghidare reținute vizează ARNm cu secvență perfect complementară și o reprimă prin scindare de proteina Ago2 la poziția nucleotidică 10 a catenei de ghidare siRNA (Elbashir și colab., 2001; Hammond și colab., 2001; Martinez și colab., 2002). Cu toate acestea, o dovadă acumulată din experimentele la nivel de genom indică faptul că un număr mare de ARNm cu complementarități parțiale la firul de ghidare sunt, de asemenea, reduse (Jackson și colab., 2003, 2006; Lim și colab., 2005; Birmingham și colab., 2006; Ui-Tei și colab., 2008). Acest fenomen este denumit efect off-țintă dependent de semințe și observat de preferință în ARNm 3′ UTRs. Mecanismul de recunoaștere a țintei acestui efect în afara țintei este cunoscut a fi similar cu cel al tăcerii genei mediate de miARN (Lewis și colab., 2005; Lim și colab., 2005; Grimson și colab., 2007). Transcrierile cu secvențe complementare regiunii de semințe poziționate 2-8 de la Terminalul 5 ‘ sunt în principal reduse. Se știe că regiunea de semințe este situată la suprafața Ago într-o formă cvasi-elicoidală pentru a servi drept loc de intrare sau nucleație pentru ARN-urile mici din risc (Ma și colab., 2005; Yuan și colab., 2005). Astfel, regiunea de semințe identifică mai întâi ARNm-urile țintă și, ulterior, formează o asociere perfectă a bazei cu ARNm țintă intenționată și induce ARNr prin Ago2.
pe baza mecanismului ARNr, se consideră că un siRNA specific genei țintă este selectabil în conformitate cu următoarele trei etape.
pasul 1: Selectarea secvenței funcționale de siARN
eficiența de eliminare a siARN se dovedește a fi foarte dependentă de secvențele lor. Am susținut regula bazată empiric care a prescris caracteristicile sirn-urilor extrem de funcționale (Ui-Tei și colab., 2004), o astfel de regulă se numește regula Ui-Tei (Figura 1). SiRNA selectat de regula Ui-Tei îndeplinesc simultan următoarele patru condiții: (1) A sau U la poziția 1 de la capătul de 5′ al firului de ghidare a siARN, (2) G sau C la poziția 19, (3) au bogăție (au 4) în pozițiile 1-7 și (4) nu se întinde GC lung 10. Cu excepția (4), regula noastră a indicat că siARN funcțional are stabilitate asimetrică în terminalele 5′ și 3′. Validarea noastră experimentală utilizând testul luciferase reporter a arătat că 98% dintre sirn-urile care îndeplinesc condițiile de mai sus au redus expresia luciferase reporter sub 33% (Ui-Tei și colab., 2004). Alte grupuri au demonstrat, de asemenea, regulile sirn-urilor extrem de funcționale denumite regula Reynolds (Reynolds și colab., 2004) și regula Amarzguioui (Amarzguioui și Prydz, 2004) așa cum sunt rezumate în Figura 1. Aceste reguli au arătat, de asemenea, în mod clar că sirn-urile funcționale sunt asimetrice: o catenă ARN cu terminal instabil de 5′ a fost eficientă ca o catenă de ghidare. Mai mult decât atât, comun în aceste reguli, 5′ terminus al firului de ghidare siRNA funcțional a fost de preferat să fie A sau U. s-a dezvăluit mai târziu că rezultatele au reflectat caracteristicile structurale ale ago2 umane (Frank și colab., 2010). Structura cristalină a unui domeniu mediu (mediu) din experimentele de titrare ago2 și RMN umane a arătat că monofosfații nucleotidici, AMP și UMP, se leagă cu o afinitate de până la 30 de ori mai mare decât CMP sau GMP, oferind dovezi structurale pentru interacțiunile specifice nucleotidelor în domeniul mediu al proteinelor eucariote în urmă.
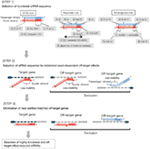
Figura 1. Reprezentare schematică pentru selectarea efectului funcțional și off-țintă-siRNAs reduse. Selectarea siRNA extrem de funcțional de Ui-Tei, Reynolds, reguli Amarzguioui, sau o combinație a acestora (pasul 1). Selectarea siRNAs cu stabilitate scăzută în duplexurile țintă de semințe (pasul 2). Eliminarea siRNAs cu secvențe potrivite aproape perfecte cu gene non-țintă (Pasul 3). În fiecare regulă, poziția nucleotidelor indică numărul de nucleotide numărate de la Terminalul 5 ‘ al catenei de ghidare.  :
:  A/U în poziția 1.
A/U în poziția 1.  G / C la poziția 19.
G / C la poziția 19.  4 până la 7 A/SUA în pozițiile 1-7.
4 până la 7 A/SUA în pozițiile 1-7. 
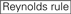 :
:  conținut de GC (30% -52%).
conținut de GC (30% -52%).  a/u 3 la pozițiile 1-5.
a/u 3 la pozițiile 1-5.  absența repetărilor interne.
absența repetărilor interne.  A în poziția 1.
A în poziția 1.  A la poziția 17.
A la poziția 17.  U în poziția 10.
U în poziția 10.  Nu G / C la poziția 1.
Nu G / C la poziția 1.  Nu G la poziția 7.
Nu G la poziția 7. 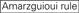 :
:  o asimetrie în stabilitatea capetelor duplex (măsurată ca diferențialul a/U al celor trei perechi de bază terminale la fiecare capăt al duplexului).
o asimetrie în stabilitatea capetelor duplex (măsurată ca diferențialul a/U al celor trei perechi de bază terminale la fiecare capăt al duplexului).  G sau C la poziția 19.
G sau C la poziția 19.  A sau U la poziția 1.
A sau U la poziția 1.  A sau U la poziția 14.
A sau U la poziția 14.  nu U la poziția 19.
nu U la poziția 19.  Nu G în poziția 1.
Nu G în poziția 1.
pasul 2: Selectarea secvenței siARN cu efecte Off-țintă reduse
pentru a evita efectele off-țintă dependente de semințe, o abordare poate fi selectarea catenei de ghidare a siARN a cărei secvență de semințe nu este complementară niciunei secvențe din 3′ UTR a tuturor genelor nețintă. Cu toate acestea, această abordare s-a dovedit a fi imposibilă, deoarece sirn-urile umane cu cea mai rară secvență de semințe de șapte nt aveau încă complementarități de semințe cu mai multe ARNm ne-vizate. Deci, am căutat regulile care guvernează capacitatea sirn-urilor de a induce efectul off-țintă dependent de semințe și am arătat că eficiența efectului off-țintă este foarte corelată cu stabilitatea termodinamică a duplexului format între regiunea de semințe a firului de ghidare siRNA și ARNm-ul său țintă (Ui-Tei și colab., 2008). Temperatura de topire( Tm), unul dintre parametrii termodinamici pentru formarea duplexului ARN, a arătat o corelație pozitivă puternică cu inducerea efectelor off-țintă dependente de semințe. Astfel, selectarea sirn-urilor cu Tm scăzut al duplexului țintă-semințe ar trebui să reducă la minimum tăcerea off-țintă dependentă de semințe (Figura 1). Tm – ul de 21,5 CTC poate servi drept punct de referință, care diferențiază secvențele de semințe aproape fără țintă de cele pozitive în afara țintei. În plus, deoarece efectul în afara țintei poate fi cauzat nu numai de firul de ghidare, ci și de firul pasager, sirn-urile al căror Tm țintă de semințe este suficient de scăzut pentru ambele fire sunt favorabile.
Pasul 3: Eliminarea genelor Off-țintă aproape perfecte potrivite
chiar și atunci când valoarea Tm a duplexului țintă-sămânță este suficient de scăzută, tăcerea genei țintă poate avea loc în continuare dacă Regiunea non-sămânță este complet complementară. Prin urmare, în a treia etapă, sirn-urile care au potriviri aproape perfecte cu orice alte transcrieri ne-vizate au fost eliminate (Figura 1).
software de proiectare siARN
am prezentat software-ul de proiectare siARN, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figura 2), care oferă software funcțional, specific țintă de proiectare a siARN în conformitate cu procedurile menționate mai sus (Naito și colab., 2009). În parametrul implicit, siRNAs satisface regula Ui-Tei pot fi selectate. Atunci când sirn-urile funcționale candidate ar putea forma duplexuri țintă-semințe cu valori Tm sub 21,5 CTC, iar regiunile lor de 19 nt care acoperă pozițiile 2-20 din ambele fire au cel puțin două neconcordanțe cu orice alte transcrieri ne-vizate, siDirect 2.0 poate proiecta cel puțin un siARN calificat pentru >94% din secvențele ARNm umane din RefSeq.

Figura 2. Vizualizări ecran de siDirect 2.0 siRNA software-ul de proiectare. (A) pagina de sus (http://siDirect2.RNAi.jp/). (B) parametri opționali pentru proiectarea siARN. (C) pagina de rezultate. (D) lista detaliată a candidaților în afara țintei cu potriviri aproape perfecte. Alinierea dintre fiecare transcriere în afara țintei și secvența siRNA vizualizează pozițiile nepotrivirilor.
alte programe software pentru selectarea sirn-urilor funcționale au fost deschise publicului așa cum se arată în tabelul 1. În multe dintre acestea, regula Ui-Tei (Ui-Tei și colab., 2004), Reynolds rule (Reynolds și colab., 2004), regula Amarzguioui (Amarzguioui și Prydz, 2004), regula Tuschl (Elbashir și colab., 2002), iar combinația acestora a fost frecvent și larg utilizată. Pentru a elimina genele non-țintă potrivite aproape perfecte, BLAST search a fost utilizat pentru căutarea omologiei în mai multe software-uri. Cu toate acestea, deoarece BLAST search nu este atât de precis pentru secvențe scurte precum siRNAs, siDirect, WU-BLAST și Bowtie, care sunt foarte precise motor de căutare omologie pentru secvențe scurte sunt adesea folosite. Dintre acestea, siDirect 2.0 poate aduce cele mai precise rezultate. În plus, unele dintre software-ul ia în considerare caracteristicile suplimentare, cum ar fi structura secundară ARNm (Ladunga, 2007; Lu și Mathews, 2008), despicare alternativă (Park și colab., 2008) sau secvența motivului prin care ar putea apărea răspunsul imun al unui virus ARN (Gong și colab., 2008). Aceste caracteristici nu sunt luate în considerare în siDirect 2.0. Deci, pentru a lua în considerare aceste caracteristici, sirna-urile selectate în mod obișnuit de siDirect 2.0 și celelalte programe software adecvate pot produce rezultate optime.
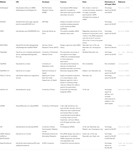
Tabelul 1. Programe software mici de proiectare a ARN-ului interferent.
în plus, este practic important să se utilizeze două sau mai multe ARNm care vizează site-uri diferite într-o genă țintă intenționată, deoarece efectele de eliminare ale unei gene țintă intenționate ar trebui să fie comune, dar efectele în afara țintei sunt susceptibile de a fi diferite între ARNm.
concluzie
se știe că eficacitatea fiecărui siARN variază foarte mult în funcție de secvența sa în celulele mamiferelor și doar o fracțiune limitată de siARN proiectate aleatoriu este funcțională. Mai mult, efectele de tăcere în afara țintei apar atunci când siARN are complementaritate parțială în regiunea semințelor cu gene neintenționate. Aici, pe baza mașinilor RNAi, am descris proiectarea rațională a sirn-urilor funcționale, cu efect redus în afara țintei, care se așteaptă să distrugă o genă țintă-în mod specific.
Declarație privind conflictul de interese
autorii declară că cercetarea a fost realizată în absența oricăror relații comerciale sau financiare care ar putea fi interpretate ca un potențial conflict de interese.
mulțumiri
această lucrare a fost parțial susținută de subvenții de la Ministerul Educației, Culturii, Sportului, științei și Tehnologiei din Japonia (MEXT) și proiectul de inovare celulară (MEXT) și proiectul de cercetare de bază pentru Universitatea privată; potrivirea subvenției Fondului pentru Kumiko Ui-Tei.
Amarzguioui, M. și Prydz, H. (2004). Un algoritm pentru selectarea secvențelor funcționale siRNA. Biochem. Biophys. Res. Commun. 316, 1050–1058.
Pubmed Rezumat / Pubmed Text Complet / CrossRef Text Complet
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S. și Khvorova, A. (2006). Meciurile de semințe 3 ‘ UTR, dar nu și identitatea generală, sunt asociate cu rnai în afara țintelor. Nat. Metode 3, 199-204.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Castanotto, D. și Rossi, J. J. (2009). Promisiunile și capcanele terapiei bazate pe interferențe ARN. Natura 457, 426-433.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Chalk, A. M. și Sonnhammer ,E. L. L. (2008). căutarea specificității siARN care încorporează date de toleranță la nepotrivire. Bioinformatica 24, 1316-1317.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Cui, W., Ning, J., Naik, U. P. și Duncan ,M. K. (2004). OptiRNAi, și rnai instrument de proiectare. Calcul. Metode Programe Biomed. 75, 67–73.
Pubmed Rezumat / Pubmed Text Complet / CrossRef Text Complet
Ding, Y., Chan, C. Y., și Lawrence, C. E. (2004). Server web Sfold pentru plierea statistică și proiectarea rațională a acizilor nucleici. Acizi Nucleici Res. 32, W135-W141.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K. și Tuschl, T. (2001). Duplexurile ARN-urilor 21-nucleotidice mediază interferența ARN în celulele de mamifere cultivate. Natură 411, 494-498.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Elbashir, W. M., Harborth, J., Weber, K. și Tuschl, T. (2002). Analiza funcției genice în celulele mamiferelor somatice folosind ARN-uri mici interferente. Metode 26, 199-213.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Frank, F., Sonenberg, N. și Nagar, B. (2010). Baza structurală pentru recunoașterea specifică a bazei 5 ‘ -nucleotidice a ARN-ului ghid de către ago uman2. Natură 465, 818-822.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S. și Li, Tongbin. (2008). siDRM: și eficient și în general aplicabile on-line siARN instrument de proiectare. Bioinformatica 24, 2405-2406.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P. și Bartel, D. P. (2007). Specificitatea de direcționare a microARN la mamifere: determinanți dincolo de împerecherea semințelor. Mol. Celula 27, 91-105.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R. și Hannon, G. J. (2001). Argonaute 2 legătură între analizele genetice și biochimice ale Arnai. Știință 293, 1146-1150.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Henschel, A., Buchholz, F. și Habermann, B. (2004). DEQOR: un instrument bazat pe web pentru proiectarea și controlul calității siRNAs. Acizi Nucleici Res. 32, W113-W120.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Horn, T., Sandmann, T. și Boutros, M. (2010). Proiectarea și evaluarea bibliotecilor la nivel de genom pentru ecranele de interferență ARN. Biol Genomului. 11, R61.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Jackson, A. L., Bartz, S. R., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G. și Linsley, P. S. (2003). Profilarea expresiei dezvăluie reglarea genelor în afara țintei de către RNAi. Nat. Biotehnol. 21, 635–637.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L. și Linsley, P. S. (2006). Syrna pe scară largă” off-target ” transcript tăcere mediată de complementaritatea secvenței regiunii semințelor. ARN 12, 1179-1187.
Pubmed Rezumat / Pubmed Text Complet / CrossRef Text Complet
Ketting, R. F. (2011). Multe fețe ale lui RNAi. Dev. Celula 15, 148-161.
CrossRef text complet
Khvorova, A., Reynolds, A. și Jayasena ,S. D. (2003). SiRNAs funcționale și miRNAs prezintă părtinire strand. Celula 115, 209-216.
Pubmed Rezumat / Pubmed Text Complet / CrossRef Text Complet
Ladunga, I. (2007). Reducerea mai completă a genelor de către mai puține siRNAs: design optimizat transparent și semnătură biofizică. Acizi Nucleici Res. 35, 433-440.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Leuschner, P. J., Ameres, S. L., Kueng, S. și Martinez, J. (2006). Scindarea catenei pasagerului siRNA în timpul asamblării RISC în celulele umane. EMBO Rep. 7, 314-320.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Lewis, B. P., Burge, C. B. și Bartel, D. P. (2005). Împerecherea conservată a semințelor, adesea flancată de adenozine, indică faptul că mii de gene umane sunt ținte microARN. Celula 120, 15-20.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S. și Johnson, J. M. (2005). Analiza Microarray arată că unele microARN-uri reglează în jos un număr mare de ARNm țintă. Natură 433, 769-773.
Pubmed Rezumat / Pubmed Text Complet / CrossRef Text Complet
Lu, Z. J., și Mathews, D. H. (2008). Selecție eficientă a siARN utilizând termodinamica de hibridizare. Acid Nucleic Res. 36, 640-647.
CrossRef text complet
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T. și Patel, D. J. (2005). Baza structurală pentru recunoașterea specifică 5 ‘ a ARN-ului ghid de către proteina A. fulgidus piwi. Natură 434, 666-670.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R. și Tuschl, T. (2002). SiRNAs antisens monocatenar ghidează scindarea ARN țintă în RNAi. Celula 110, 563-574.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P. și Zamore, P. D. (2005). Clivajul catenei pasagerului facilitează asamblarea siARN în complexe enzimatice RNAi care conțin Ago2. Celula 123, 607-620.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Naito, Y., Yoshimura, J., Morishita, S. și Ui-Tei, K. (2009). siDirect 2.0: software actualizat pentru proiectarea siARN funcțional cu efect off-țintă redus dependent de semințe. BMC Bioinformatica 10, 392.
CrossRef text complet
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M. și Kim, Y. J. (2008). AsiDesigner: server de proiectare siRNA bazat pe exon, având în vedere îmbinarea alternativă. Acid Nucleic Res. 36, W97-W103.
CrossRef text complet
Rand, T. A., Petersen, S., Du, F. și Wang, X. (2005). Argonaute2 scindează firul anti-ghid al siRNA în timpul activării RISC. Celula 123, 621-629.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S. și Khvorova, A. (2004). Proiectare rațională a siARN pentru interferența ARN. Nat. Biotehnol. 22, 326–330.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N. și Zamore, P. D. (2003). Asimetria în ansamblul complexului enzimatic RNAi. Celula 115, 199-208.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Shah, J. K., Garner, H. R., White, M. A., Shames, D. S. și Minna, J. D. (2007). sIR: resursă informațională siRNA, un instrument bazat pe web pentru proiectarea și analiza secvenței siRNA și o bază de date cu acces deschis siRNA. BMC Bioinformatica 8, 178. doi:10.1186/1471-2105-8-178
CrossRef text complet
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A. și Saigo, K. (2008). Stabilitatea termodinamică și asocierea bazei Watoson-Crick în duplexul semințelor sunt factori determinanți majori ai eficienței efectului off-țintă bazat pe sRNA. Acizi Nucleici Res. 36, 7100-7109.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. și Saigo, K. (2004). Linii directoare pentru selectarea secvențelor de siARN extrem de eficiente pentru interferența ARN a mamiferelor și a puilor. Acizi Nucleici Res. 32, 936-948.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Vert, J.-P., Foveau, N., Lajaunie, C. și Vandenbrouck, Y. (2006). Un model precis și interpretabil pentru predicția eficacității siARN. BMC Bioinformatica 7, 520. doi:10.1186/1471-2105-7-520
CrossRef text complet
Yuan, B., Latek, R., Rossbach, M., Tuschl, T. și Lewitter, F. (2004). server de selecție siRAN: un server automat de predicție a oligonucleotidelor siRNA. Acizi Nucleici Res. 32, W130-W134.
Pubmed rezumat / Pubmed text complet / CrossRef text complet
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T. și Patel, D. J. (2005). Structura cristalină a A. aeolicus argonaute, o endoribonuclează ghidată de ADN specifică site-ului, oferă informații despre scindarea ARNm mediată de RISC. Mol. Celula 19, 405-419.
Pubmed Rezumat / Pubmed Text Complet / CrossRef Text Complet