Úvod
RNA interference (RNAi) je široce používaná technika, kterou malé interferující RNA (siRNA) downreguluje konkrétní cílový gen, s perfektní komplementární sekvenci, a slíbil, že použití v terapeutické aplikace na lidských onemocnění (Castanotto a Rossi, 2009; Ketting, 2011). Přestože má člověk více než 20 000 genů, je žádoucí použít siRNA, která je vysoce funkční a nemá žádné účinky na jiné geny než na svůj specifický cíl. V tomto článku přezkoumáváme optimalizovanou metodu návrhu siRNA založenou na mechanismu RNAi. Kromě toho představujeme webové stránky otevřené veřejnosti pro výběr sekvencí siRNA.
duplexů 21-nukleotidové (nt) RNA s 2 NT 3′ převisy (siRNA) se obvykle používá pro experimenty RNAi. Po dodání do buněk jsou siRNA začleněny do komplexu tlumení indukovaného RNA (RISC) jako dvouvláknová RNA. RISC je efektorový komplex obsahující Argonautový protein (Ago)s aktivitou kráječe (Hammond et al., 2001; Martinez et al., 2002). SiRNA průvodce pramen obsahující termodynamicky méně stabilní 5′-konci je přednostně zachována RISC (Khvorova et al., 2003; Schwarz a kol., 2003; Ui-Tei et al., 2004). Řetězce cestujících většiny dvouvláknových siRNA naložených na RISC jsou štěpeny proteinem Ago2 a degradovány (Matranga et al ., 2005; Rand a kol., 2005; Leuschner a kol., 2006). Nerozdělený guide strand párů cílové mRNA s dokonale komplementární sekvenci, a potlačuje to tím, že štěpení Ago2 proteinu v nukleotidové pozici 10 siRNA guide strand (Elbashir et al., 2001; Hammond a kol., 2001; Martinez et al., 2002). Nicméně, nahromaděné důkazy z genomu-široký experimenty ukazují, že velký počet mRNAs s částečnou návaznost na vodící vlákna jsou také sníženy (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham a kol., 2006; Ui-Tei a kol., 2008). Tento jev je označován jako účinek mimo cíl závislý na semenech a s výhodou pozorován u mRNA 3 ‚ UTRs. Je známo, že mechanismus rozpoznávání cíle tohoto off-cílového účinku je podobný mechanismu umlčení genů zprostředkovaných miRNA (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Transkripty se sekvencemi komplementárními k oblasti semen umístěné 2-8 od 5 ‚ terminálu jsou převážně redukovány. Je známo, že oblast semen se nachází na povrchu Ago v kvazi-Šroubovité formě, která slouží jako vstupní nebo nukleační místo pro malé RNA v RISCs (Ma et al ., 2005; Yuan et al., 2005). Oblast osiva tedy nejprve identifikuje cílové mRNA a následně vytvoří dokonalé párování bází se zamýšlenou cílovou mRNA a indukuje RNAi Ago2.
na základě mechanismu RNAi se cílová genově specifická siRNA považuje za volitelnou podle následujících tří kroků.
Krok 1: Výběr funkční sekvence siRNA
účinnost knockdownu siRNA je vysoce závislá na jejich sekvencích. Obhajovali jsme empiricky založené pravidlo, které předepisovalo vlastnosti vysoce funkčních siRNA (Ui-Tei et al., 2004), takové pravidlo se nazývá pravidlo Ui-Tei (Obrázek 1). SiRNA zvolená pravidlem Ui-Tei splňuje současně následující čtyři podmínky: (1) nebo U v pozici 1 od 5′ konci siRNA guide strand, (2) G nebo C, na pozici 19, (3) AU bohatství (AU ≥4) v pozice 1-7, a (4) žádné dlouhé GC úsek ≥10. S výjimkou (4) naše pravidlo ukázalo, že funkční siRNA má asymetrickou stabilitu v 5′ a 3 ‚ svorkách. Naše experimentální validace pomocí luciferase reporter assay ukázala, že 98% siRNA splňujících výše uvedené podmínky snížilo expresi luciferase reporter pod 33% (Ui-Tei et al ., 2004). Jiné skupiny také prokázaly pravidla vysoce funkčních siRNA označovaných jako Reynoldsovo pravidlo (Reynolds et al., 2004) a amarzguioui rule (Amarzguioui and Prydz, 2004) shrnuto na obrázku 1. Tato pravidla také jasně ukázala, že funkční siRNA jsou asymetrické: řetězec RNA s nestabilním 5 ‚ terminálem byl účinný jako vodicí řetězec. Kromě toho, časté v těchto pravidlech, 5‘ konci funkční siRNA průvodce pramen byl lepší než být nebo U. To bylo později ukázalo, že výsledky odrážejí strukturální rysy lidské Ago2 (Frank et al., 2010). Krystalová struktura MID (střední) domény z lidské AGO2 a NMR titrační experimenty ukázaly, že nukleotid monophosphates, AMP a UMP, svázat s až 30-krát vyšší afinitu než buď CMP nebo GMP, zajištění strukturální důkazy pro nukleotid-specifické interakce v POLOVINĚ domény eukaryotických PŘED bílkovin.
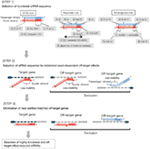
Obrázek 1. Schematické znázornění pro výběr funkční a off-target efekt-snížení sirna. Výběr vysoce funkční siRNA podle Ui-Tei, Reynolds, amarzguioui pravidel, nebo jejich kombinace (Krok 1). Výběr siRNA s nízkou stabilitou v duplexech osiva a cíle (krok 2). Eliminace siRNA s téměř dokonalými sladěnými sekvencemi s necílovými geny (Krok 3). V každém pravidle Poloha nukleotidu udává počet nukleotidů počítaných od 5 ‚ terminálu vodícího řetězce.  :
:  A / U na pozici 1.
A / U na pozici 1.  G / C na pozici 19.
G / C na pozici 19.  4 až 7 A / Us v pozicích 1-7.
4 až 7 A / Us v pozicích 1-7. 
 :
:  obsah GC (30% -52%).
obsah GC (30% -52%).  A / U ≥ 3 v pozicích 1-5.
A / U ≥ 3 v pozicích 1-5.  Absence vnitřních opakování.
Absence vnitřních opakování.  A na pozici 1.
A na pozici 1.  A na pozici 17.
A na pozici 17.  U na pozici 10.
U na pozici 10.  není G / C na pozici 1.
není G / C na pozici 1.  není G na pozici 7.
není G na pozici 7. 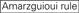 :
:  asymetrie stability duplexních konců (měřeno jako A / U diferenciál tří koncových párů na obou koncích duplexu).
asymetrie stability duplexních konců (měřeno jako A / U diferenciál tří koncových párů na obou koncích duplexu).  G nebo C na pozici 19.
G nebo C na pozici 19.  A nebo U na pozici 1.
A nebo U na pozici 1.  A nebo U na pozici 14.
A nebo U na pozici 14.  není U na pozici 19.
není U na pozici 19.  není G na pozici 1.
není G na pozici 1.
Krok 2: Výběr siRNA Sekvence s Omezenou Off-Cíl Účinky
, Aby se zabránilo semeno-závislé off-cíl účinky, jeden přístup může být do vyberte siRNA průvodce pramen, jehož seed sekvence je komplementární k nějaké sekvence na 3′ UTR všech non-cílené geny. Tento přístup se však ukázal jako nemožný, protože lidské siRNA s nejčastější sekvencí sedmi nt semen měly stále komplementarity semen s několika necílenými mRNA. Takže, podívali jsme se na pravidla, která upravují způsobilost sirna přimět semínko-závislé off-target efekt, a ukázalo se, že účinnost off-target efekt je vysoce závislé na termodynamické stability duplex tvořil mezi semen regionu siRNA guide strand a jeho cílové mRNA (Ui-Tei et al., 2008). Teplota tání (Tm), jeden z termodynamické parametry pro tvorbu RNA duplex, ukázala silnou pozitivní korelaci s indukční osiva-závislé off-cíl účinky. Výběr siRNA s nízkou Tm duplexu osiva-cíl by tedy měl minimalizovat umlčení mimo cíl závislé na osivu (Obrázek 1). TM 21,5°C může sloužit jako měřítko, které rozlišuje sekvence semen téměř bez cíle od sekvencí pozitivních mimo cíl. Kromě toho, protože off-target efekt může být způsoben nejen tím, že průvodce pramen, ale také osobní pramen, sirna, jejichž osivo-cíl Tm je dostatečně nízké pro oba prameny jsou příznivé.
Krok 3: Odstranění téměř Perfektní Uzavřeno Off-Cílové Geny
I když Tm hodnoty osiva-cíl duplex je dostatečně nízká, cíl umlčování genů může stále dojít, pokud non-seed region je zcela komplementární. Proto ve třetím kroku byly odstraněny siRNA, které mají téměř dokonalé shody s jinými necílenými přepisy (Obrázek 1).
siRNA Design Software
představili Jsme siRNA design software, siDirect 2.0 (http://siDirect2.RNAi.jp/; Obrázek 2), který poskytuje funkční, cílově specifický návrhový software siRNA podle výše uvedených postupů (Naito et al., 2009). Ve výchozím parametru lze zvolit siRNA splňující pravidlo Ui-Tei. Když kandidát funkční sirna by mohl tvořit semeno-cíl duplexy s Tm hodnoty pod 21,5°C, a jejich 19-nt regiony zahrnující pozice 2-20 oba prameny mají alespoň dvě neshody jakékoliv jiné non-cílené přepisy, siDirect 2.0 můžete navrhnout alespoň jednu kvalifikovanou siRNA pro >94% lidské mRNA sekvence v RefSeq.

Obrázek 2. Zobrazení obrazovky siDirect 2.0 siRNA návrhového softwaru. (A) horní strana (http://siDirect2.RNAi.jp/). B) volitelné parametry pro návrh siRNA. (C) stránka s výsledky. (D) podrobný seznam kandidátů mimo cíl s téměř dokonalými zápasy. Zarovnání mezi každým off-cílovým přepisem a sekvencí siRNA vizualizuje pozice nesouladů.
jiný software pro výběr funkčních siRNA byl otevřen veřejnosti, jak ukazuje tabulka 1. V mnoha z nich pravidlo Ui-Tei (Ui-Tei et al., 2004), Reynolds rule (Reynolds et al., 2004), amarzguioui rule (Amarzguioui and Prydz, 2004), Tuschl rule (Elbashir et al., 2002) a jejich kombinace byla často a široce používána. K odstranění téměř dokonalých shodných necílových genů, BLAST search byl použit pro homologické vyhledávání v několika programech. Nicméně, protože VÝBUCH hledání není tak přesné, pro krátké sekvence, jako sirna, siDirect, WU-BLAST, a Motýlek, které jsou vysoce přesné homologie vyhledávač pro krátké sekvence jsou často používány. Mezi nimi může siDirect 2.0 přinést nejpřesnější výsledky. Kromě toho, některé z software zvážit další funkce, jako je sekundární struktura mRNA (Ladunga, 2007; Lu a Mathews, 2008), alternativní sestřih (Park et al., 2008) nebo sekvence motivů, při které může dojít k imunitní odpovědi virem RNA (Gong et al., 2008). Tyto funkce nejsou v siDirect 2.0 brány v úvahu. Aby bylo možné tyto funkce zohlednit, mohou siRNA běžně vybraná siDirect 2.0 a dalšími vhodnými softwarovými programy produkovat optimální výsledek.
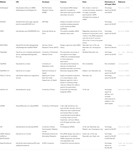
Tabulka 1. Malé interferující softwarové programy pro návrh RNA.
kromě toho, že je prakticky důležité zvážit použití dvou nebo více sirna cílení na různých místech v určených cílových genů, protože poražený účinky zamýšlený cílový gen, by měli být společné, ale off-cíl účinky jsou pravděpodobně být různé mezi sirna.
Závěr
účinnost každé siRNA je známo, že je široce liší v závislosti na jeho pořadí v savčích buňkách, a pouze zlomek z náhodně určené sirna je funkční. Kromě toho dochází k tlumení mimo cíl, když má siRNA částečnou komplementaritu v oblasti semen s nezamýšlenými geny. Tady, na základě strojů RNAi, popsali jsme racionální návrh funkčních, siRNA se sníženým efektem mimo cíl, u kterých se očekává, že srazí cílový Gen-konkrétně.
Střet Zájmů Prohlášení
autoři prohlašují, že výzkum byl prováděn v nepřítomnosti jakékoli obchodní nebo finanční vztahy, které by mohlo být chápáno jako potenciální konflikt zájmů.
Poděkování
Tato práce byla částečně podporována granty od Ministerstva Školství, Kultury, Sportu, Vědy a Technologie Japonska (MEXT), a Buňka Inovační Projekt (MEXT), a Základní Výzkum Projekt pro Soukromé Univerzitě; odpovídající fond dotaci Kumiko Ui-Tei.
Amarzguioui, M., and Prydz, h. (2004). Algoritmus pro výběr funkčních siRNA sekvencí. Biochem. Biophys. Res.Commun. 316, 1050–1058.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef Celý Text
Birmingham, A., Anderson, E. M., Reynolds, a., Ilsley-Tyree, D., Leake, D., Fedorov, y., Baskerville, s., Maksimova, e., Robinson, k., Karpilow, J., Marshall, W. S., and Khvorova, a. (2006). 3 ‚ UTR seed zápasy, ale ne celková identita, jsou spojeny s RNAi off-cíle. Adresa. Metody 3, 199-204.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Castanotto, D., a Rossi, j. J. (2009). Sliby a úskalí terapeutik založených na interferenci RNA. Příroda 457, 426-433.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Chalk, am, and Sonnhammer, E. L. L. (2008). vyhledávání specifičnosti siRNA zahrnující údaje o toleranci nesouladu. Bioinformatika 24, 1316-1317.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Cui, W., Ning, J., Naik, U. P., a Duncan, M. K. (2004). OptiRNAi, a RNAi návrhový nástroj. Comput. Metody Programy Biomed. 75, 67–73.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Ding, Y., Chan, C. Y., a Lawrence, C. E. (2004). Sfold webový server pro statistické skládání a racionální návrh nukleových kyselin. Nukleové Kyseliny Rez.32, W135–W141.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K., a Tuschl, T. (2001). Duplexy 21-nukleotidových RNA zprostředkovávají interferenci RNA v kultivovaných savčích buňkách. Příroda 411, 494-498.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Elbashir, W. M., Harborth, J., Weber, K., a Tuschl, T. (2002). Analýza genové funkce v somatických savčích buňkách pomocí malých interferujících RNA. Metody 26, 199-213.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Frank, F., Sonenberg, N., a Nagar, B. (2010). Strukturní základ pro 5′-nukleotidové báze specifické rozpoznávání vodicí RNA lidským AGO2. Příroda 465, 818-822.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Gong, W., Ren, y., Zhou, h., Wang, y., Kang, S. A Li, Tongbin. (2008). siDRM: a efektivní a obecně použitelné on-line siRNA návrhový nástroj. Bioinformatika 24, 2405-2406.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P., a Bartel, D. P. (2007). Specificita cílení na mikroRNA u savců: determinanty mimo párování semen. Molo. Cela 27, 91-105.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Hammond, S. M., Boettcher, S., Caudy, a. a., Kobayashi, R., a Hannon, G. J. (2001). Argonaute 2 vazba mezi genetickými a biochemickými analýzami RNAi. Věda 293, 1146-1150.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Henschel, a., Buchholz, F., and Habermann, B. (2004). DEQOR: webový nástroj pro návrh a kontrolu kvality siRNA. Nukleové Kyseliny Rez.32, W113–W120.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Horn, T., Sandmann, T., and Boutros, m. (2010). Návrh a vyhodnocení genomových knihoven pro RNA interferenční obrazovky. Genom Biol. 11, R61.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Jackson, A. L., Bartz, S. R., Přístřeší, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., a Linsley, P. S. (2003). Profilování exprese odhaluje regulaci genů mimo cíl pomocí RNAi. Adresa. Biotechnol. 21, 635–637.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L., a Linsley, P. S. (2006). Rozšířené tlumení transkriptu siRNA“ mimo cíl “ zprostředkované komplementaritou sekvence semen. RNA 12, 1179-1187.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Ketting, R. F. (2011). Mnoho tváří RNAi. Rozvoj. Cela 15, 148-161.
CrossRef Plný Text
Khvorova, a., Reynolds, A., and Jayasena, S. D. (2003). Funkční siRNA a Mirna vykazují zkreslení pramenů. Cela 115, 209-216.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Ladunga, I. (2007). Úplnější umlčení genů menším počtem siRNA: transparentní optimalizovaný design a biofyzikální podpis. Nukleové Kyseliny Rez.35, 433-440.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Leuschner, P. J., Ameres, S. L., Kueng, a. S., a Martinez, J. (2006). Štěpení řetězce cestujících siRNA během montáže RISC V lidských buňkách. EMBO Rep. 7, 314-320.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Lewis, B. P., Burge, C. B., a Bartel, D. P. (2005). Konzervované párování semen, často lemované adenosiny, naznačuje, že tisíce lidských genů jsou mikroRNA cíli. Cela 120, 15-20.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Zámek, J., Bartel, D. P., Linsley, P. S. a Johnson, J. M. (2005). Microarray analýza ukazuje, že některé mikroRNA downregulují velké množství cílových mRNA. Příroda 433, 769-773.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Lu, Z. J., a Mathews, D.H. (2008). Efektivní výběr siRNA pomocí hybridizační termodynamiky. Nukleová Kyselina Res.36, 640-647.
CrossRef celý Text
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T., a Patel, D. J. (2005). Strukturní základ pro 5 ‚ – end-specifické rozpoznání vodicí RNA proteinem a.fulgidus piwi. Příroda 434, 666-670.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Martinez, J., Patkaniowska, a., Urlaub, h., Luhrmann, R., and Tuschl, T. (2002). Jednovláknové antisense siRNA vedou cílové štěpení RNA v RNAi. Cela 110, 563-574.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Matranga, C., Tomari, Y. Shin, C., Bartel, D. P., a Zamore, P. D. (2005). Štěpení pasažérů usnadňuje sestavení siRNA do enzymových komplexů RNAi obsahujících Ago2. Cela 123, 607-620.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Naito, y., Yoshimura, J., Morishita, s., and Ui-Tei, k. (2009). siDirect 2.0: aktualizovaný software pro navrhování funkční siRNA se sníženým efektem off-target závislým na osivu. BMC Bioinformatics 10, 392.
CrossRef celý Text
Park, Y.-K., Park, S.-M., Choi, Y.-C. Lee, D., Získal, M., Kim, Y. J. (2008). AsiDesigner: exon-based siRNA design server zvažuje alternativní sestřih. Nukleová Kyselina Res.36, W97–W103.
CrossRef Plný Text
rand, T. a., Petersen, s., Du, F., and Wang, X. (2005). Argonaute2 štěpí anti-vodicí řetězec siRNA během aktivace RISC. Cela 123, 621-629.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Reynolds, A., Leake, D., Boese, Q, Scaringe, S., Marshall, W. S., a Khvorova, A. (2004). Racionální návrh siRNA pro interferenci RNA. Adresa. Biotechnol. 22, 326–330.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Schwarz, D. S., Hutvangner, G., Du, T, Xu, Z., Aronin, N., a Zamore, P. D. (2003). Asymetrie v sestavě enzymového komplexu RNAi. Cela 115, 199-208.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Šach, J. K., Garner, H. R., Bílý, M. a., Škoda, D. S., a Minna, J. D. (2007). siRNA information resource, webový nástroj pro návrh a analýzu sekvencí siRNA a databázi siRNA s otevřeným přístupem. BMC bioinformatika 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Plný Text
Ui-Tei, k., Naito, y., Nishi, k., Juni, A., and Saigo, k. (2008). Termodynamická stabilita a párování bází Watoson-Crick v duplexu semen jsou hlavními determinanty účinnosti off-cílového efektu založeného na sRNA. Nukleové Kyseliny Rez.36, 7100-7109.
Pubmed Abstrakt / Pubmed Plný Text / CrossRef Plný Text
Ui-Tei, k., Naito, y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, h., Juni, a., Ueda, R., and Saigo, k. (2004). Pokyny pro výběr vysoce účinných sekvencí siRNA pro interferenci savců a kuřat RNA. Nukleové Kyseliny Rez.32, 936-948.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Vert, J.-P., Foveau, N., Lajaunie, C., a Vandenbrouck, Y. (2006). Přesný a interpretovatelný model pro predikci účinnosti siRNA. BMC bioinformatika 7, 520. doi:10.1186/1471-2105-7-520
CrossRef celý Text
Juanů, B., Latek, R., Rossbach, M., Tuschl, T., a Lewitter, F. (2004). Siran selection server: automatizovaný server predikce oligonukleotidů siRNA. Nukleové Kyseliny Rez.32, W130–W134.
Pubmed Abstraktní | Pubmed Plný Text | CrossRef celý Text
Juanů, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T., a Patel, D. J. (2005). Krystalová struktura a. aeolicus argonaute, endoribonukleáza řízená site-specific DNA, poskytuje pohled na štěpení mRNA zprostředkované RISC. Molo. Cela 19, 405-419.
Pubmed Abstrakt | Pubmed Plný Text / CrossRef Plný Text