Introduction
RNA interferenssiä (RNAi) on yleisesti käytetty tekniikka, jolla small interfering RNA (siRNA) alentaa tietyn kohdegeenin säätelyä täydellisellä komplementaarisella sekvenssillä ja jota luvataan käyttää ihmisten sairauksien hoidossa (Castanotto and Rossi, 2009; Ketting, 2011). Vaikka ihmisellä on yli 20 000 geeniä, on suotavaa käyttää sirnaa, joka on erittäin toimiva ja jolla ei ole vaikutuksia muihin geeneihin kuin sen tiettyyn kohteeseen. Tässä artikkelissa tarkastelemme optimoitua menetelmää sirnan suunnittelemiseksi RNAi: n mekanismin perusteella. Lisäksi otamme käyttöön yleisölle avoimet verkkosivut siRNA-sekvenssien valitsemiseksi.
Sirnan optimoitu rakenne
Duplexes of 21-nukleotidi (nt) RNA with 2 nt 3′ overhangs (siRNA) käytetään yleensä RNAi-kokeissa. Soluihin siirryttäessä sirnat liitetään RNA: n indusoimaan hiljentämiskompleksiin (RISC) kaksijuosteisena RNA: na. RISC on efektorikompleksi, joka sisältää Argonaute-proteiinia (Ago), jolla on slicer-aktiivisuus (Hammond et al., 2001; Martinez et al., 2002). SiRNA-ohjainjuonne, joka sisältää termodynaamisesti vähemmän stabiilin 5′ – pään, säilyy ensisijaisesti RISC: llä (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). Useimpien RISC:hen lastattujen kaksijuosteisten sirnojen matkustajasäikeet halkaistaan Ago2-proteiinilla ja hajoavat (Matranga et al., 2005; Rand et al., 2005; Leuschner ym., 2006). Jäljellä olevat ohjainjuosteparit kohdistavat mRNA: n täydellisesti toisiaan täydentävään sekvenssiin ja tukahduttavat sen pilkkoutumalla Ago2-proteiinilla siRNA-ohjainjuosteen nukleotidiasemassa 10 (Elbashir et al., 2001; Hammond ym., 2001; Martinez ym., 2002). Genomin laajuisista kokeista kertynyt näyttö osoittaa kuitenkin, että suuri määrä mrnoja, joilla on osittainen täydentävyys ohjenuoraan, vähenee myös (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham ym., 2006; Ui-Tei et al., 2008). Tätä ilmiötä kutsutaan siemenriippuvaiseksi kohteen ulkopuoliseksi vaikutukseksi, ja se havaitaan mieluiten mRNA 3′ UTRs-järjestelmässä. Tämän off-target-vaikutuksen kohdetunnistusmekanismin tiedetään olevan samanlainen kuin miRNA-välitteisen geenin hiljentämisen (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Transkriptit, joiden sekvenssit täydentävät siemenaluetta, joka on sijoitettu 2-8 5 ’ – päätteestä, vähenevät pääasiassa. Siemenalueen tiedetään sijaitsevan Agon pinnalla kvasikierteisessä muodossa, joka toimii pienten RNA: iden sisäänmeno-tai nukleaatiopaikkana Risceissä (Ma et al., 2005; Yuan ym., 2005). Näin siemenalue tunnistaa ensin kohdealueen mrnat ja muodostaa sen jälkeen täydellisen emäsparinmuodostuksen aiotun kohdealueen mRNA: n kanssa ja indusoi RNAi: n ago2: lla.
RNAi: n mekanismin perusteella kohdegeenispesifinen siRNA katsotaan valittavaksi seuraavien kolmen vaiheen mukaan.
Vaihe 1: funktionaalisen siRNA-sekvenssin valinta
sirnojen knockdown-tehokkuus paljastuu hyvin riippuvaiseksi niiden sekvensseistä. Kannatimme empiirisesti perustuvaa sääntöä, joka määräsi erittäin toiminnallisten sirnojen ominaisuudet (Ui-Tei et al., 2004), tällaista sääntöä kutsutaan Ui-Tei-säännöksi (Kuva 1). Ui-Tei-säännön valitsema siRNA täyttää seuraavat neljä ehtoa samanaikaisesti: (1) A tai U kohdassa 1 sirnan ohjainjuoman 5′ terminuksesta, (2) G tai C kohdassa 19, (3) au: n rikkaus (AU ≥4) kohdissa 1-7 ja (4) pitkä GC-venytys ≥10. Lukuun ottamatta (4) sääntömme osoitti, että funktionaalisessa sirnassa on epäsymmetrinen vakaus 5′ ja 3′ – terminaaleissa. Kokeellinen validointimme käyttäen luciferase reporter-määritystä osoitti, että 98% edellä mainitut edellytykset täyttävistä sirnoista vähensi luciferase Reporterin ilmentymistä alle 33%: iin(Ui-Tei et al., 2004). Muut ryhmät osoittivat myös erittäin funktionaalisten sirnojen säännöt, joihin viitataan nimellä Reynoldsin sääntö (Reynolds et al., 2004) ja Amarzguioui rule (Amarzguioui and Prydz, 2004), kuten on tiivistetty kuviossa 1. Nämä säännöt osoittivat myös selvästi, että toiminnalliset sirnat ovat epäsymmetrisiä: RNA-juoste, jossa on epävakaa 5′ – pääte, oli tehokas ohjainjuosteena. Lisäksi yleinen näissä säännöissä, 5 ’ terminus funktionaalisen siRNA guide strand oli parempi olla A tai U. myöhemmin paljastui, että tulokset heijastivat rakenteellisia piirteitä ihmisen Ago2 (Frank et al., 2010). Ihmisen AGO2-ja NMR-titrauskokeista saatu keskikohdan (middle) kiderakenne osoitti, että nukleotidimonofosfaatit AMP ja UMP sitoutuvat jopa 30 kertaa suuremmalla affiniteetilla kuin joko CMP tai GMP, mikä antaa rakenteellista näyttöä nukleotidispesifisistä vuorovaikutuksista eukaryoottisten AGO-proteiinien KESKIKOHTEESSA.
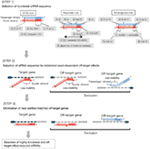
Kuva 1. Kaavamainen esitys toiminnallisten ja off-target vaikutus-vähennetty siRNAs. Valinta erittäin toimiva siRNA Ui-Tei, Reynolds, Amarzguioui säännöt, tai niiden yhdistelmä (Vaihe 1). Valikoima sirnoja, joiden stabiilisuus siemenkohteessa on alhainen (Vaihe 2). Sirnojen eliminointi lähes täydellisillä sekvensseillä ei-kohdegeeneihin (Vaihe 3). Kussakin säännössä nukleotidiasento ilmaisee, kuinka monta nukleotidia lasketaan ohjainjuosteen 5′ – terminaalista. 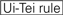 :
:  A / U sija 1.
A / U sija 1.  G / C sijalla 19.
G / C sijalla 19.  4-7 a / Us sijoilla 1-7.
4-7 a / Us sijoilla 1-7. 
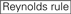 :
:  GC-pitoisuus (30% – 52%).
GC-pitoisuus (30% – 52%).  A / U ≥ 3 kohdissa 1-5.
A / U ≥ 3 kohdissa 1-5.  sisäisten toistojen puuttuminen.
sisäisten toistojen puuttuminen.  a sijalla 1.
a sijalla 1.  a sijalla 17.
a sijalla 17.  U sijalla 10.
U sijalla 10.  ei G/C asemassa 1.
ei G/C asemassa 1. 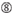 ei G sijalla 7.
ei G sijalla 7. 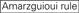 :
:  duplex-päiden stabiilisuuden epäsymmetria (mitattuna kolmen päätepohjan a/U-differentiaalina dupleksin kummassakin päässä).
duplex-päiden stabiilisuuden epäsymmetria (mitattuna kolmen päätepohjan a/U-differentiaalina dupleksin kummassakin päässä).  G tai C sijalla 19.
G tai C sijalla 19.  A tai U sija 1.
A tai U sija 1.  A tai U sijalla 14.
A tai U sijalla 14.  ei U sijalla 19.
ei U sijalla 19.  ei G sijalla 1.
ei G sijalla 1.
Vaihe 2: SiRNA-sekvenssin valinta, jolla on vähäisemmät Off-Target-vaikutukset
, jotta vältetään siemenriippuvaiset off-target-vaikutukset, yksi lähestymistapa voi olla valita siRNA-ohjelohko, jonka siemenjärjestys ei täydennä mitään sekvenssejä kaikkien ei-kohdennettujen geenien 3′ UTR: ssä. Tämä lähestymistapa on kuitenkin osoittautunut mahdottomaksi, koska ihmisen sirnoilla, joilla oli harvinaisin seitsenkertainen siemenjärjestys, oli edelleen siementäydennyksiä useiden Ei-kohdennettujen mrnojen kanssa. Olemme siis etsineet sääntöjä, jotka säätelevät sirnojen kykyä aiheuttaa siemenriippuvaista off-target-vaikutusta, ja paljastaneet, että off-target-vaikutuksen tehokkuus korreloi suuresti siRNA guide Strandin siemenalueen ja sen kohteen mRNA: n välille muodostuvan dupleksin termodynaamiseen stabiilisuuteen (Ui-Tei et al., 2008). Sulamislämpötila (TM), yksi RNA-dupleksin muodostumisen termodynaamisista parametreista, osoitti voimakasta positiivista korrelaatiota siemenriippuvaisten off-target-vaikutusten induktion kanssa. Näin ollen valitsemalla sirnat, joilla on alhainen TM siemenkohteessa, olisi minimoitava siemenriippuvainen kohteen ulkopuolinen äänenvaimennus (Kuva 1). Vertailukohtana voi olla 21,5°C: n Tm, joka erottaa lähes kohdepositiiviset siemenjaksot muista kuin kohdepositiivisista. Lisäksi koska kohteen ulkopuolinen vaikutus voi johtua paitsi ohjainjuovasta myös matkustajajuovasta, sirnat, joiden siemenkohtainen Tm on riittävän alhainen molemmille säikeille, ovat suotuisia.
Vaihe 3: Lähes täydellisten Matched Off-Target-geenien
eliminoituminen silloinkin, kun siemen-kohde-dupleksin TM-arvo on riittävän alhainen, kohdegeenin hiljentyminen voi silti tapahtua, jos muu kuin siemenalue on täysin toisiaan täydentävä. Siksi kolmannessa vaiheessa sirnat, joilla on lähes täydellinen vastaavuus muiden ei-kohdennettujen transkriptien kanssa, eliminoitiin (Kuva 1).
siRNA Design Software
esitimme siRNA design software, siDirect 2.0 (http://siDirect2.RNAi.jp/; Kuva 2), joka tarjoaa toiminnallisia, kohdekohtaisia siRNA-suunnitteluohjelmia edellä mainittujen menettelyjen mukaisesti (Naito et al., 2009). Oletusparametrissa voidaan valita Sirnas täyttävän Ui-Tei-säännön. Kun kandidaatin funktionaaliset sirnat voivat muodostaa siemenkohtaisia duplexeja, joiden Tm-arvo on alle 21,5°C, ja niiden 19-nt-alueet, jotka ulottuvat molempien säikeiden asemiin 2-20, ovat vähintään kaksi yhteensopimatonta muiden ei-kohdennettujen transkriptien kanssa, siDirect 2.0 voi suunnitella vähintään yhden pätevän sirnan >94%: lle ihmisen mRNA-sekvensseistä RefSeq: ssa.

kuva 2. Näytönäkymät siDirect 2.0 siRNA design-ohjelmistosta. A) yläsivu (http://siDirect2.RNAi.jp/). (B) Sirna designin valinnaiset parametrit. C) tulossivu. (D) yksityiskohtainen luettelo paitsioehdokkaista, joilla on lähes täydelliset osumat. Kohdistus kunkin off-target transkription ja siRNA-sekvenssin välillä visualisoi kohtausvirheiden sijainnit.
muut ohjelmistot toiminnallisten sirnojen valitsemiseksi olivat avoinna yleisölle taulukon 1 mukaisesti. Monissa niistä Ui-Tei sääntö (Ui-Tei et al., 2004), Reynoldsin sääntö (Reynolds et al., 2004), Amarzguiouin sääntö (Amarzguioui and Prydz, 2004), Tuschlin sääntö (Elbashir et al., 2002), ja niiden yhdistelmä oli usein ja laajalti käytetty. Lähes täydellisesti sovitettujen ei-kohdegeenien eliminoimiseksi BLAST search-sovellusta käytettiin homologisessa etsinnässä useissa ohjelmissa. Koska BLAST search ei kuitenkaan ole niin tarkka lyhyille sekvensseille kuten siRNAs, siDirect, WU-BLAST ja Bowtie, jotka ovat erittäin tarkkoja homologiahakukoneita lyhyille sekvensseille käytetään usein. Niistä siDirect 2.0 saattaa tuoda tarkimmat tulokset. Lisäksi osa ohjelmistoista ottaa huomioon lisäominaisuudet, kuten mRNA-sekundaarirakenne (Ladunga, 2007; Lu and Mathews, 2008), vaihtoehtoiset liitokset (Park et al., 2008)tai motif-sekvenssi, jolla RNA-viruksen aiheuttama immuunivaste voi tapahtua (Gong et al., 2008). Näitä ominaisuuksia ei huomioida siDirect 2.0: ssa. Jotta nämä ominaisuudet voitaisiin ottaa huomioon, siDirect 2.0: n yleisesti valitsemat sirnat ja muut sopivat ohjelmat voivat tuottaa optimaalisen tuloksen.
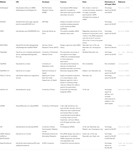
Taulukko 1. Pieniä häiritseviä RNA-suunnitteluohjelmia.
lisäksi on käytännössä tärkeää käyttää kahta tai useampaa sirnaa, jotka kohdistuvat aiotun kohdegeenin eri kohtiin, koska aiotun kohdegeenin knockdown-vaikutusten oletetaan olevan yleisiä, mutta kohteen ulkopuoliset vaikutukset ovat todennäköisesti erilaisia sirnojen välillä.
johtopäätös
kunkin sirnan tehon tiedetään vaihtelevan suuresti riippuen sen sekvenssistä nisäkässoluissa, ja vain pieni osa satunnaisesti suunnitelluista sirnoista on toiminnallisia. Lisäksi kohteen ulkopuolisia hiljentymisvaikutuksia syntyy, kun sirnalla on siemenalueella osittainen täydentävyys tahattomien geenien kanssa. Tässä, perustuu RNAi koneet, kuvasimme rationaalinen suunnittelu toiminnallisia, off-target vaikutus-vähennetty siRNAs, joiden odotetaan tyrmätä kohdegeeni-erityisesti.
Eturistiriitalausunto
kirjoittajat toteavat, että tutkimus tehtiin ilman kaupallisia tai taloudellisia suhteita, joita voitaisiin pitää mahdollisena eturistiriitana.
kiitokset
tätä työtä tuettiin osittain Japanin opetus -, kulttuuri -, urheilu -, tiede-ja teknologiaministeriön (MEXT), cell Innovation Projectin (MEXT) ja yksityisen yliopiston Ydintutkimushankkeen myöntämillä avustuksilla.
Amarzguioui, M., and Prydz, H. (2004). Algoritmi funktionaalisten siRNA-jaksojen valintaan. Biochem. Biofyysejä. Res. Commun. 316, 1050–1058.
Pubmed Abstrakti / Pubmed Koko Teksti | CrossRef Koko Teksti
Birmingham, A., Anderson, Em., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S., and Khvorova, A. (2006). 3 ’ UTR seed matches, mutta ei yleistä identiteettiä, liittyy RNAi off-tavoitteet. Nat. Menetelmät 3, 199-204.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Castanotto, D., and Rossi, J. J. (2009). RNA-interferenssipohjaisen terapeutiikan lupaukset ja sudenkuopat. Nature 457, 426-433.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Chalk, A. M., and Sonnhammer, E. L. L. (2008). siRNA spesifisyys haku sisältää epäsuhta toleranssi tiedot. Bioinformatiikka 24, 1316-1317.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
cui, W., Ning, J., Naik, U. P., and Duncan, M. K. (2004). OptiRNAi, ja RNAi suunnittelutyökalu. Comput. Menetelmät Ohjelmat Biomed. 75, 67–73.
Pubmed Abstrakti / Pubmed Koko Teksti | CrossRef Koko Teksti
Ding, Y., Chan, C. Y., ja Lawrence, 2004). Sfold web-palvelin tilastollinen taitto ja järkevä suunnittelu nukleiinihappojen. Nukleiinihapot Res. 32, W135–W141.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K., and Tuschl, T. (2001). 21-nukleotidin RNA-välitteiset dupleksit välittävät RNA-häiriöitä viljellyissä nisäkässoluissa. Nature 411, 494-498.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Elbashir, W. M., Harborth, J., Weber, K., and Tuschl, T. (2002). Geenin toiminnan analysointi somaattisissa nisäkässoluissa käyttäen pieniä häiritseviä RNA: ita. Methods 26, 199-213.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Frank, F., Sonenberg, N., and Nagar, B. (2010). Rakenteellinen perusta 5 ’ -nukleotidiemäksispesifiselle opas-RNA: n tunnistamiselle ihmisen AGO2: n avulla. Nature 465, 818-822.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S. ja Li, Tongbin. (2008). siDRM: sekä tehokas ja yleisesti sovellettavissa oleva siRNA-suunnittelutyökalu verkossa. Bioinformatiikka 24, 2405-2406.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P., and Bartel, D. P. (2007). MicroRNA targeting specificity in mammals: determinants beyond seed parinmuodostus. Mol. Selli 27, 91-105.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R., and Hannon, G. J. (2001). Argonaute 2 linkki RNAi: n geneettisten ja biokemiallisten analyysien välillä. Science 293, 1146-1150.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Henschel, A., Buchholz, F., and Habermann, B. (2004). DEQOR: web-pohjainen työkalu sirnasin suunnitteluun ja laadunvalvontaan. Nukleiinihapot Res. 32, W113–W120.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Horn, T., Sandmann, T., and Boutros, M. (2010). Genominlaajuisten kirjastojen suunnittelu ja arviointi RNA-interferenssinäytöille. Genomibiolia. 11, R61.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Jackson, A. L., Bartz, S. R., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., and Linsley, P. S. (2003). Expression profilointi paljastaa off-target geenin sääntelyn RNAi. Nat. Bioteknologiaa. 21, 635–637.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L., and Linsley, P. S. (2006). Laaja siRNA ”off-target” transkriptio hiljentäminen välittämä siemenalueen sekvenssin täydentävyys. RNA 12, 1179-1187.
Pubmed Abstrakti / Pubmed Koko Teksti | CrossRef Koko Teksti
Ketting, R. F. (2011). Rnain monet kasvot. Dev. Selli 15, 148-161.
CrossRef koko teksti
Khvorova, A., Reynolds, A., and Jayasena, S. D. (2003). Toiminnalliset sirnat ja mirnat näytteille strand bias. Selli 115, 209-216.
Pubmed Abstrakti / Pubmed Koko Teksti | CrossRef Koko Teksti
Ladunga, I. (2007). Täydellisempi geenien hiljentäminen vähemmällä sirnalla: läpinäkyvä optimoitu muotoilu ja biofyysinen allekirjoitus. Nucleic Acids Res. 35, 433-440.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Leuschner, P. J., Ameres, S. L., Kueng, S., and Martinez, J. (2006). SiRNA-matkustajajuoman pilkkoutuminen RISC-kokoonpanon aikana ihmissoluissa. EMBO Rep. 7, 314-320.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Lewis, B. P., Burge, C. B., and Bartel, D. P. (2005). Säilöttyjen siementen pariutuminen, jota adenosiinit usein reunustavat, osoittaa, että tuhannet ihmisen geenit ovat mikroRNA-kohteita. Selli 120, 15-20.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Lim, LP, Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S., and Johnson, J. M. (2005). Mikroarray analyysi osoittaa, että jotkut microRNAs downregulate suuri määrä target mRNAs. Nature 433, 769-773.
Pubmed Abstrakti / Pubmed Koko Teksti | CrossRef Koko Teksti
Lu, ZJ., ja Mathews, D. H. (2008). Tehokas siRNA-valinta hybridisaatiotermodynamiikan avulla. Nucleic Acid Res. 36, 640-647.
CrossRef koko teksti
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T., and Patel, D. J. (2005). Rakenteellinen perusta 5 ’ -endispesifiselle opas-RNA: n tunnistamiselle A. fulgidus piwi-proteiinin avulla. Nature 434, 666-670.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R., and Tuschl, T. (2002). Yksisäikeinen antisense siRNAs ohjaa kohde-RNA: n pilkkoutumista RNAi: ssa. Selli 110, 563-574.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., and Zamore, P. D. (2005). Passenger-strand pilkkominen helpottaa siRNA: n yhdistämistä Ago2: ta sisältäviksi RNAi-entsyymikomplekseiksi. Selli 123, 607-620.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Naito, Y., Yoshimura, J., Morishita, S., and Ui-Tei, K. (2009). siDirect 2.0: päivitetty ohjelmisto funktionaalisen sirnan suunnitteluun, jossa on pienempi siemenriippuvainen off-target-vaikutus. BMC Bioinformatics 10, 392.
CrossRef koko teksti
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M., and Kim, Y. J. (2008). AsiDesigner: exon-pohjainen siRNA-suunnittelupalvelin harkitsee vaihtoehtoista liittämistä. Nucleic Acid Res. 36, W97-W103.
CrossRef koko teksti
Rand, T. A., Petersen, S., Du, F., and Wang, X. (2005). Argonaute2 katkaisee sirnan anti-guide-juosteen RISC-aktivoinnin aikana. Selli 123, 621-629.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S., and Khvorova, A. (2004). Rationaalinen siRNA-suunnittelu RNA-interferenssille. Nat. Bioteknologiaa. 22, 326–330.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N., and Zamore, P. D. (2003). Epäsymmetria RNAi-entsyymikompleksin kokoonpanossa. Selli 115, 199-208.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Shah, J. K., Garner, H. R., White, M. A., Shames, D. S., and Minna, J. D. (2007). sirna information resource, web-pohjainen työkalu siRNA sekvenssin suunnitteluun ja analysointiin sekä avoin siRNA-tietokanta. BMC Bioinformatics 8, 178. doi:10.1186/1471-2105-8-178
yliviivataan koko teksti
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A., and Saigo, K. (2008). Termodynaaminen Stabiilisuus ja Watoson-Crick – emäsparitus siemendupleksissa ovat merkittäviä sRNA-pohjaisen off-target-vaikutuksen tehokkuutta määrittäviä tekijöitä. Nucleic Acids Res. 36, 7100-7109.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R., and Saigo, K. (2004). Ohjeet erittäin tehokkaiden siRNA-sekvenssien valitsemiseksi nisäkäs-ja Tipu-RNA-interferensseihin. Nucleic Acids Res. 32, 936-948.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Vert, J.-P., Foveau, N., Lajaunie, C., and Vandenbrouck, Y. (2006). Tarkka ja tulkittava malli sirnan tehokkuuden ennustamiseen. BMC Bioinformatics 7, 520. doi:10.1186/1471-2105-7-520
yliviivataan koko teksti
Yuan, B., Latek, R., Rossbach, M., Tuschl, T., and Lewitter, F. (2004). siRAN selection server: automaattinen siRNA-oligonukleotidien ennustuspalvelin. Nukleiinihapot Res. 32, W130–W134.
Pubmed Abstrakti / Pubmed koko teksti | CrossRef koko teksti
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T., and Patel, D. J. (2005). A. aeolicus argonauten kiderakenne, paikkasidonnainen DNA-ohjattu endoribonukleaasi, antaa oivalluksia RISC-välitteisestä mRNA-pilkkoutumisesta. Mol. Selli 19, 405-419.
Pubmed Abstrakti / Pubmed Kokoteksti / CrossRef Kokoteksti