- Introduzione
- Design ottimizzato di siRNA
- FASE 1: Selezione della sequenza siRNA funzionale
- FASE 2: Selezione della sequenza siRNA con effetti off-target ridotti
- FASE 3: Eliminazione dei geni Off-Target quasi perfetti
- siRNA Software di progettazione
- Conclusione
- Dichiarazione sul conflitto di interessi
- Riconoscimenti
Introduzione
interferenza del RNA (RNAi) è ampiamente utilizzato una tecnica di piccolo RNA interferente (siRNA) sottoregola uno specifico gene bersaglio con una perfetta sequenza complementare, e ha promesso di utilizzare in applicazioni terapeutiche per malattie umane (Castanotto e Rossi, 2009; Ketting, 2011). Sebbene l’essere umano abbia più di 20.000 geni, è desiderabile usare siRNA che è altamente funzionale e non ha effetti su alcun gene diverso dal suo obiettivo specifico. In questo articolo, esaminiamo un metodo ottimizzato per progettare siRNA basato sul meccanismo di RNAi. Inoltre, introduciamo i siti web aperti al pubblico per la selezione di sequenze siRNA.
Design ottimizzato di siRNA
Duplex di 21-nucleotide (nt) RNA con 2 nt 3′ sporgenze (siRNA) è di solito utilizzato per esperimenti RNAi. Al momento della consegna nelle cellule, i SIRNA sono incorporati nel complesso di silenziamento indotto dall’RNA (RISC) come RNA a doppio filamento. RISC è il complesso effettuatore contenente proteina Argonaute (Fa) con attività affettatrice (Hammond et al., 2001; Martinez et al., 2002). Il filo guida siRNA contenente l’estremità 5’termodinamicamente meno stabile è preferenzialmente mantenuto da RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). I fili passeggeri della maggior parte dei SIRNA a doppio filamento caricati su RISC sono scissi dalla proteina Ago2 e degradati (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). Le coppie di filamenti guida trattenute mirano all’mRNA con una sequenza perfettamente complementare e lo reprimono per scissione dalla proteina Ago2 in posizione nucleotidica 10 del filo guida siRNA (Elbashir et al., 2001; Hammond et al., 2001; Martinez et al., 2002). Tuttavia, una prova accumulata da esperimenti su tutto il genoma indica che un gran numero di MRNA con complementarità parziali al filo guida sono anche ridotti (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Questo fenomeno è indicato come effetto fuori bersaglio dipendente dal seme e preferibilmente osservato in mRNA 3 ‘ UTRs. Il meccanismo di riconoscimento target di questo effetto off-target è noto per essere simile a quello del silenziamento genico mediato da miRNA (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). I trascritti con sequenze complementari alla regione del seme posizionata 2-8 dal 5’ terminale sono principalmente ridotti. La regione del seme è nota per essere situata sulla superficie di Ago in una forma quasi elicoidale per servire come sito di ingresso o nucleazione per piccoli RNA nei RISC(Ma et al., 2005; Yuan et al., 2005). Così, la regione del seme in primo luogo identifica gli mRNA dell’obiettivo e successivamente forma l’base-accoppiamento perfetto con l’mRNA dell’obiettivo destinato ed induce RNAi da Ago2.
Sulla base del meccanismo di RNAi, un siRNA gene-specifico dell’obiettivo è considerato selezionabile secondo i seguenti tre punti.
FASE 1: Selezione della sequenza siRNA funzionale
Le efficienze di knockdown dei SIRNA sono rivelate altamente dipendenti dalle loro sequenze. Abbiamo sostenuto la regola basata empiricamente che prescriveva le caratteristiche dei SIRNA altamente funzionali (Ui-Tei et al., 2004), tale regola è chiamata come regola Ui-Tei (Figura 1). Il siRNA selezionato da Ui-Tei regola soddisfare le seguenti quattro condizioni contemporaneamente: (1) A o U in posizione 1 da 5′ estremità del filo guida siRNA, (2) G o C in posizione 19, (3) AU ricchezza (AU ≥4) in posizioni 1-7, e (4) nessun lungo GC stretch ≥10. Fatta eccezione per (4), la nostra regola indicava che il siRNA funzionale ha stabilità asimmetrica nei terminali 5′ e 3′. La nostra convalida sperimentale utilizzando il test luciferase reporter ha mostrato che il 98% dei SIRNA che soddisfano le condizioni di cui sopra ha ridotto l’espressione di luciferase reporter al di sotto del 33% (Ui-Tei et al., 2004). Altri gruppi hanno anche dimostrato le regole dei siRNA altamente funzionali indicati come regola di Reynolds (Reynolds et al., 2004) e Amarzguioui rule (Amarzguioui e Prydz, 2004) come riassunto nella Figura 1. Queste regole hanno anche mostrato chiaramente che i SIRNA funzionali sono asimmetrici: un filamento di RNA con terminale 5′ instabile era efficace come filo guida. Inoltre, comune in queste regole, 5 ‘terminus di funzionale siRNA filo guida era preferibile essere A o U. E’ stato rivelato in seguito che i risultati riflettevano le caratteristiche strutturali di Ago2 umana (Frank et al., 2010). La struttura cristallina di un dominio MEDIO (medio) da esperimenti di titolazione AGO2 e NMR umani ha mostrato che i monofosfati nucleotidici, AMP e UMP, si legano con un’affinità fino a 30 volte superiore rispetto a CMP o GMP, fornendo prove strutturali per le interazioni specifiche dei nucleotidi nel dominio MEDIO delle proteine FA eucariotiche.
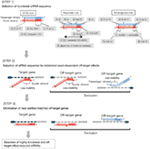
Figura 1. Rappresentazione schematica per la selezione di SIRNA funzionali e off-target effetto ridotto. Selezione di siRNA altamente funzionale da Ui-Tei, Reynolds, Amarzguioui regole, o la loro combinazione (FASE 1). Selezione di SIRNA con bassa stabilità nei duplex semi-bersaglio (FASE 2). Eliminazione di SIRNA con sequenze quasi perfette abbinate a geni non bersaglio (FASE 3). In ogni regola, la posizione del nucleotide indica il numero di nucleotidi contati dal terminale 5′ del filo guida. 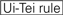 :
:  A / U alla posizione 1.
A / U alla posizione 1.  G / C alla posizione 19.
G / C alla posizione 19.  da 4 a 7 A / Us nelle posizioni 1-7.
da 4 a 7 A / Us nelle posizioni 1-7. 
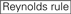 :
:  Contenuto GC (30% -52%).
Contenuto GC (30% -52%).  A/U ≥ 3 alle posizioni 1-5.
A/U ≥ 3 alle posizioni 1-5.  Assenza di ripetizioni interne.
Assenza di ripetizioni interne.  A alla posizione 1.
A alla posizione 1.  A alla posizione 17.
A alla posizione 17.  U alla posizione 10.
U alla posizione 10.  Non G / C alla posizione 1.
Non G / C alla posizione 1.  Non G alla posizione 7.
Non G alla posizione 7. 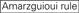 :
:  Un’asimmetria nella stabilità delle estremità duplex (misurata come differenziale A / U delle tre coppie di base terminali alle due estremità del duplex).
Un’asimmetria nella stabilità delle estremità duplex (misurata come differenziale A / U delle tre coppie di base terminali alle due estremità del duplex).  G o C alla posizione 19.
G o C alla posizione 19.  A o U alla posizione 1.
A o U alla posizione 1.  A o U alla posizione 14.
A o U alla posizione 14.  Non U alla posizione 19.
Non U alla posizione 19.  Non G alla posizione 1.
Non G alla posizione 1.
FASE 2: Selezione della sequenza siRNA con effetti off-target ridotti
Per evitare effetti off-target dipendenti dal seme, un approccio può essere quello di selezionare il filo guida siRNA la cui sequenza di semi non è complementare a nessuna sequenza nel 3′ UTR di tutti i geni non mirati. Tuttavia, questo approccio si è dimostrato impossibile, perché i SIRNA umani con la sequenza di semi sette-nt più rara avevano ancora complementarità di semi con diversi MRNA non mirati. Quindi, abbiamo cercato le regole che governano la capacità dei siRNA di indurre un effetto off-target dipendente dal seme e abbiamo rivelato che l’efficienza dell’effetto off-target è altamente correlata alla stabilità termodinamica del duplex formato tra la regione del seme del filo guida siRNA e il suo mRNA target (Ui-Tei et al., 2008). La temperatura di fusione (Tm), uno dei parametri termodinamici per la formazione di RNA duplex, ha mostrato una forte correlazione positiva con l’induzione di effetti off-target dipendenti dal seme. Pertanto, la selezione dei SIRNA con Tm bassa del duplex seed-target dovrebbe ridurre al minimo il silenziamento off-target dipendente dal seme (Figura 1). Il Tm di 21,5°C può servire come punto di riferimento, che discrimina le sequenze del seme quasi fuori bersaglio-libere da quelle fuori bersaglio-positive. Inoltre, poiché l’effetto fuori bersaglio può essere causato non solo dal filo guida ma anche dal filo passeggero, i SIRNA il cui Tm bersaglio seme è sufficientemente basso per entrambi i fili sono favorevoli.
FASE 3: Eliminazione dei geni Off-Target quasi perfetti
Anche quando il valore Tm del duplex seed-target è sufficientemente basso, il silenziamento genico target può ancora avvenire se la regione non-seed è completamente complementare. Pertanto, nella terza fase, i SIRNA che hanno corrispondenze quasi perfette con qualsiasi altra trascrizione non mirata sono stati eliminati (Figura 1).
siRNA Software di progettazione
Abbiamo presentato siRNA software di progettazione, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figura 2), che fornisce un software di progettazione siRNA funzionale e specifico per il target secondo le procedure sopra menzionate (Naito et al., 2009). Nel parametro predefinito, è possibile selezionare siRNAs che soddisfa la regola Ui-Tei. Quando i SIRNA funzionali candidati potrebbero formare duplex seed-target con valori Tm inferiori a 21,5°C e le loro regioni 19-nt che coprono le posizioni 2-20 di entrambi i trefoli hanno almeno due disallineamenti a qualsiasi altro trascritto non mirato, siDirect 2.0 può progettare almeno un siRNA qualificato per >94% delle sequenze mRNA umane in RefSeq.

Figura 2. Viste sullo schermo del software di progettazione siRNA siDirect 2.0. (A) Pagina principale (http://siDirect2.RNAi.jp/). (B) Parametri facoltativi per progettazione di siRNA. (C) Pagina dei risultati. (D) Elenco dettagliato dei candidati fuori bersaglio con partite quasi perfette. L’allineamento tra ogni trascrizione off-target e la sequenza siRNA visualizza le posizioni di disallineamenti.
Altri software per selezionare SIRNA funzionali erano aperti al pubblico come mostrato nella Tabella 1. In molti di questi, Ui-Tei regola (Ui-Tei et al., 2004), Reynolds rule (Reynolds et al., 2004), regola di Amarzguioui (Amarzguioui e Prydz, 2004), Regola di Tuschl (Elbashir et al., 2002), e la loro combinazione è stata frequentemente e ampiamente utilizzata. Per eliminare i geni non bersaglio abbinati quasi perfetti, BLAST search è stato utilizzato per la ricerca di omologia in diversi software. Tuttavia, dal momento che la ricerca BLAST non è così preciso per brevi sequenze come siRNAs, siDirect, WU-BLAST, e Bowtie, che sono motore di ricerca di omologia altamente accurata per brevi sequenze sono spesso utilizzati. Tra questi, siDirect 2.0 può portare i risultati più accurati. Inoltre, alcuni software considerano le funzionalità aggiuntive, come mRNA secondary structure (Ladunga, 2007; Lu e Mathews, 2008), alternative splicing (Park et al., 2008), o la sequenza di motivi con cui potrebbe verificarsi la risposta immunitaria da parte di un virus a RNA (Gong et al., 2008). Queste caratteristiche non sono considerate in siDirect 2.0. Quindi, per tenere conto di queste caratteristiche, siRNAs comunemente selezionati da siDirect 2.0 e gli altri programmi software appropriati possono produrre risultati ottimali.
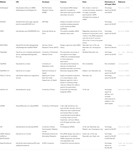
Tabella 1. Piccoli programmi software di progettazione RNA interferenti.
Inoltre, è praticamente importante utilizzare due o più SIRNA che mirano a siti diversi in un gene target previsto, poiché gli effetti di knockdown di un gene target previsto dovrebbero essere comuni, ma è probabile che gli effetti fuori bersaglio siano diversi tra i SIRNA.
Conclusione
È noto che l’efficacia di ciascun siRNA varia ampiamente a seconda della sua sequenza nelle cellule di mammifero e solo una frazione limitata di SIRNA progettati in modo casuale è funzionale. Inoltre, gli effetti di silenziamento off-target sorgono quando il siRNA ha una parziale complementarità nella regione del seme con i geni non intenzionali. Qui, sulla base dei macchinari RNAi, abbiamo descritto la progettazione razionale di SIRNA funzionali e con effetto ridotto, che dovrebbero abbattere un gene bersaglio, in particolare.
Dichiarazione sul conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di rapporti commerciali o finanziari che potrebbero essere interpretati come un potenziale conflitto di interessi.
Riconoscimenti
Questo lavoro è stato parzialmente sostenuto da sovvenzioni del Ministero dell’Istruzione, della Cultura, dello sport, della Scienza e della Tecnologia del Giappone (MEXT), e dal progetto di innovazione cellulare (MEXT), e dal progetto di ricerca principale per l’università privata; sovvenzione del fondo corrispondente a Kumiko Ui-Tei.
Amarzguioui, M., e Prydz, H. (2004). Un algoritmo per la selezione di sequenze siRNA funzionali. Biochimica. Biophys. Res. Commun. 316, 1050–1058.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Birmingham, A., Anderson, E. M. Nel 2006, Ilsley-Tyree, Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S., e Khvorova, A. (2006). 3 ‘ partite seme UTR, ma non l’identità complessiva, sono associati con RNAi off-obiettivi. NAT. Metodi 3, 199-204.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Castanotto, D., e Rossi, J. J. (2009). Le promesse e le insidie delle terapie basate sull’interferenza dell’RNA. Natura 457, 426-433.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Chalk, A. M., e Sonnhammer, E. L. L. (2008). Ricerca di specificità siRNA che incorpora dati di tolleranza di mancata corrispondenza. Bioinformatica 24, 1316-1317.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Il suo nome deriva da quello di un uomo, un uomo, un uomo e una donna. OptiRNAi, e RNAi strumento di progettazione. Calcolo. Metodi Programmi Biomed. 75, 67–73.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Ding, Y., Chan, C. Y., e Lawrence, E. V. (2004). Sfold web server per il folding statistico e la progettazione razionale degli acidi nucleici. Acidi nucleici Res. 32, W135–W141.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K. e Tuschl, T. (2001). I duplex di RNA a 21 nucleotidi mediano l’interferenza dell’RNA nelle cellule di mammifero coltivate. Natura 411, 494-498.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Elbashir, WM, Harborth, J., Weber, K. e Tuschl, T. (2002). Analisi della funzione genica in cellule somatiche di mammifero utilizzando piccoli RNA interferenti. Metodi 26, 199-213.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2010 è stato pubblicato il primo album in studio della band. Base strutturale per il riconoscimento specifico della base nucleotidica 5’dell’RNA guida da parte dell’AGO2 umano. Natura 465, 818-822.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S., e Li, Tongbin. (2008). siDRM: e strumento di progettazione siRNA online efficace e generalmente applicabile. Bioinformatica 24, 2405-2406.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2007 è stato pubblicato il primo album in studio della cantante, “The World”, pubblicato nel 2007. microRNA targeting specificità nei mammiferi: determinanti al di là di accoppiamento seme. Mol. Cella 27, 91-105.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2001 è stato pubblicato il primo album in studio della band, “The World”, pubblicato nel 2001. Argonaute 2 collegamento tra analisi genetiche e biochimiche di RNAi. Scienza 293, 1146-1150.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Henschel, A., Buchholz, F., e Habermann, B. (2004). DEQOR: uno strumento web-based per la progettazione e il controllo qualità di siRNAs. Acidi nucleici Res. 32, W113–W120.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Horn, T., Sandmann, T., e Boutros, M. (2010). Progettazione e valutazione di librerie genome-wide per schermi di interferenza RNA. Genome Biol. 11, R61.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2003 è stato pubblicato il primo album in studio della band, “The World”, pubblicato nel 2003. La profilazione di espressione rivela la regolazione del gene off-target da parte di RNAi. NAT. Biotecnologia. 21, 635–637.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2006 è stato pubblicato il primo album in studio del gruppo musicale statunitense The Rock, pubblicato nel 2006. Trascrizione diffusa siRNA” off-target ” silenziamento mediata dalla complementarità sequenza regione seme. RNA 12, 1179-1187.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Ketting, R. F. (2011). I tanti volti di RNAi. Dev. Cella 15, 148-161.
Testo integrale CrossRef
Khvorova, A., Reynolds, A., e Jayasena, S. D. (2003). I SIRNA funzionali e i miRNA presentano una distorsione del filo. Cella 115, 209-216.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Ladunga, I. (2007). Silenziamento genico più completo con un minor numero di SIRNA: design ottimizzato trasparente e firma biofisica. Acidi nucleici Res. 35, 433-440.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Leuschner, PJ, Ameres, SL, Kueng, S., e Martinez, J. (2006). Scissione del filamento passeggero siRNA durante l’assemblaggio RISC in cellule umane. EMBO Rep. 7, 314-320.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Il suo nome deriva dal greco antico, che significa “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra”, “terra” e “terra”. L’accoppiamento di semi conservati, spesso affiancato da adenosina, indica che migliaia di geni umani sono bersagli di microRNA. Cella 120, 15-20.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2005 è stato pubblicato il primo album in studio della band, “The World”, pubblicato nel 2005. L’analisi dei microarray mostra che alcuni microRNA regulano un gran numero di MRNA target. Natura 433, 769-773.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Lu, ZJ., e Mathews, D. H. (2008). Efficiente selezione siRNA utilizzando termodinamica ibridazione. Acido nucleico Res. 36, 640-647.
Testo integrale CrossRef
Ma, J.-B., Yuan, YR, Meister, G., Pei, Y., Tuschl, T., e Patel, DJ (2005). Base strutturale per il riconoscimento 5 ‘ – end-specifico dell’RNA guida da parte della proteina A. fulgidus piwi. Natura 434, 666-670.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R., e Tuschl, T. (2002). Singolo filamento antisenso siRNAs guida bersaglio RNA scissione in RNAi. Cella 110, 563-574.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Matranga, C., Tomari, Y., Shin, C., Bartel, PD, e Zamore, PD (2005). La scissione del Passeggero-filo facilita l’assemblea di siRNA nei complessi enzimatici contenenti Ago2 RNAi. Cella 123, 607-620.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2009 è stato pubblicato il primo album in studio della cantante. siDirect 2.0: software aggiornato per la progettazione di siRNA funzionale con effetto off-target semi-dipendente ridotto. BMC Bioinformatica 10, 392.
Testo integrale CrossRef
Nel 2008 è stato pubblicato il primo album in studio della cantante. AsiDesigner: server di progettazione siRNA basato su esone che considera lo splicing alternativo. Acido nucleico Res. 36, W97–W103.
Testo integrale CrossRef
Rand, TA, Petersen, S., Du, F., e Wang, X. (2005). Argonaute2 fende il filo anti-guida di siRNA durante l’attivazione RISC. Cella 123, 621-629.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2004 è stato pubblicato il primo album in studio del gruppo musicale statunitense The Mighty, pubblicato nel 2004. Progettazione razionale di siRNA per interferenza del RNA. NAT. Biotecnologia. 22, 326–330.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Schwarz, DS, Hutvangner, G., Du, T., Xu, Z., Aronin, N., e Zamore, PD (2003). Asimmetria nell’assemblaggio del complesso enzimatico RNAi. Cella 115, 199-208.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Shah, JK, Garner, HR, White, M. A., Shames, DS, e Minna, JD (2007). sIR: siRNA information resource, uno strumento basato sul Web per la progettazione e l’analisi delle sequenze siRNA e un database siRNA ad accesso aperto. BMC Bioinformatica 8, 178. doi:10.1186/1471-2105-8-178
Testo integrale CrossRef
Nel 2008 è stato pubblicato il primo album in studio della cantante. La stabilità termodinamica e l’accoppiamento della base di Watoson-Crick nel duplex del seme sono determinanti importanti dell’efficienza dell’effetto fuori bersaglio basato su sRNA. Acidi nucleici Res. 36, 7100-7109.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2004 è stato pubblicato il primo album in studio del gruppo musicale statunitense Ueda, pubblicato nel 2004. Linee guida per la selezione di sequenze siRNA altamente efficaci per l’interferenza di RNA di mammiferi e pulcini. Acidi nucleici Res. 32, 936-948.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2006 è stato pubblicato il primo album in studio della band. Un modello accurato ed interpretabile per la previsione di efficacia di siRNA. BMC Bioinformatica 7, 520. doi:10.1186/1471-2105-7-520
Testo integrale CrossRef
Yuan, B., Latek, R., Rossbach, M., Tuschl, T., e Lewitter, F. (2004). siRAN selection server: un server di previsione siRNA oligonucleotide automatizzato. Acidi nucleici Res. 32, W130–W134.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text
Nel 2005 è stato pubblicato il primo album in studio della band, “The World”, pubblicato nel 2005. La struttura cristallina di A. aeolicus argonaute, una endoribonucleasi guidata dal DNA sito-specifico, fornisce informazioni sulla scissione dell’mRNA mediata dal RISC. Mol. Cella 19, 405-419.
Pubmed Abstract / Pubmed Full Text / CrossRef Full Text