Innledning
RNA interferens (rnai) er et bredt brukt teknikk der små forstyrrende RNA (siRNA) downregulates et bestemt mål gen med perfekt komplementær sekvens, og lovet å bruke i terapeutisk anvendelse for menneskelige sykdommer (Castanotto Og Rossi, 2009; Ketting, 2011). Selv om mennesket har mer enn 20.000 gener, er det ønskelig å bruke siRNA som er svært funksjonell og har ingen effekter på andre gener enn sitt spesifikke mål. I denne artikkelen vurderer vi en optimalisert metode for å designe siRNA basert På mekanismen Til RNAi. I tillegg introduserer vi nettstedene som er åpne for publikum for å velge siRNA-sekvenser.
Optimalisert Design av siRNA
Duplekser av 21-nukleotid (nt) RNA med 2 nt 3 ‘ overheng (siRNA) brukes vanligvis Til rnai-eksperimenter. Ved levering inn i cellene blir siRNAs innlemmet I RNA-indusert silencing complex (RISC) som et dobbeltstrenget RNA. RISC er effektorkomplekset som inneholder Argonaute protein (Ago) med sliceraktivitet (Hammond et al., 2001; Martinez et al., 2002). SiRNA guide tråd som inneholder thermodynamically mindre stabil 5 ‘ – end er fortrinnsvis beholdt AV RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). Passasjerstrenger av de fleste av de dobbeltstrengede siRNAs lastet på RISC spaltes Av Ago2 protein og degradert (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). De beholdt guide tråd parene målrette mRNA med perfekt komplementær sekvens, og undertrykker den ved spalting Av Ago2 protein ved nukleotid posisjon 10 av siRNA guide strand (Elbashir et al., 2001; Hammond et al., 2001; Martinez et al., 2002). Et akkumulert bevis fra genom-brede eksperimenter indikerer imidlertid at et stort antall mrna med delvis komplementariteter til styrestrengen også reduseres (Jackson et al., 2003, 2006; Lim et al., 2005; Oslo et al., 2006; Ui-Tei et al., 2008). Dette fenomenet refereres til som frøavhengig off-target-effekt og observeres fortrinnsvis i mRNA 3 ‘ UTRs. Målgjenkjenningsmekanismen for denne off-target-effekten er kjent for å være lik den for miRNA-mediert gendeaktivering (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Transkripsjonene med sekvenser som er komplementære til frøområdet plassert 2-8 fra 5 ‘ – terminalen, reduseres hovedsakelig. Frøområdet er kjent for å ligge På Overflaten Av Ago i en kvasi-spiralformet form for å tjene som inngangssted eller kjerneområde for små Rna i Riscene (Ma et al ., 2005; Yuan et al., 2005). Dermed, frø regionen først identifiserer målet mrna, og deretter danne perfekt base-sammenkobling med tiltenkte målet mRNA og indusere RNAi Av Ago2.
basert På mekanismen Til RNAi, anses en målgen-spesifikk siRNA å være valgbar i henhold til de følgende tre trinnene.
TRINN 1: Valg Av Funksjonell siRNA Sekvens
knockdown effektiviteten av siRNAs er avslørt å være svært avhengig av deres sekvenser. Vi orde for empirisk basert regel som foreskrev egenskapene til svært funksjonelle siRNAs(Ui-Tei et al., 2004), slik regel kalles Som Ui – tei rule (Figur 1). SiRNA valgt Av Ui – tei-regelen oppfyller følgende fire betingelser samtidig: (1) a eller U i posisjon 1 fra 5 ‘ endestasjon av siRNA guide strand, (2) G eller C i posisjon 19, (3) AU rikdom (AU ≥4) i posisjoner 1-7, og (4) ingen lang gc strekning ≥10. Bortsett fra (4) indikerte vår regel at den funksjonelle siRNA har asymmetrisk stabilitet i 5′ og 3’ terminaler. Vår eksperimentelle validering ved hjelp av luciferase reporter analyse viste at 98% av siRNAs tilfredsstillende ovenfor forhold redusert uttrykk for luciferase reporter under 33% (Ui-Tei et al ., 2004). Andre grupper demonstrerte også reglene for svært funksjonelle siRNAs referert Til Som Reynolds rule (Reynolds et al., 2004) Og Amarzguioui rule (Amarzguioui Og Prydz, 2004) som oppsummert I Figur 1. Disse reglene viste også tydelig at funksjonelle sirnaer er asymmetriske: EN RNA-streng med ustabil 5 ‘ terminal var effektiv som en styrestreng. Videre vanlig i disse reglene, 5 ‘ terminus av funksjonell siRNA guide strand var å foretrekke å være en Eller U. det ble avslørt senere at resultatene reflekterte de strukturelle trekk ved human Ago2 (Frank et al ., 2010). Krystallstrukturen til ET mid (middle) domene fra humane AGO2-og NMR-titreringsforsøk viste at nukleotidmonofosfater, AMP og UMP, binder seg med opptil 30 ganger høyere affinitet enn ENTEN CMP eller GMP, noe som gir strukturelle bevis for nukleotidspesifikke interaksjoner i MID-domenet til eukaryote AGO-proteiner.
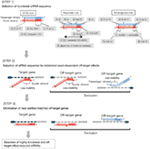
Figur 1. Skjematisk fremstilling for valg av funksjonelle og off-target effekt-redusert siRNAs. Valg av svært funksjonell siRNA Av Ui-Tei, Reynolds, Amarzguioui regler, ELLER kombinasjonen av dem (TRINN 1). Valg av siRNAs med lav stabilitet i seed-target duplexes (TRINN 2). Eliminering av siRNAs med nesten perfekte matchede sekvenser til ikke-målgener (TRINN 3). I hver regel angir nukleotidposisjonen antall nukleotid talt fra 5 ‘ terminal av styrestrengen.  :
:  A / U i posisjon 1.
A / U i posisjon 1.  G / C i posisjon 19.
G / C i posisjon 19.  4 til 7 A / Us i posisjoner 1-7.
4 til 7 A / Us i posisjoner 1-7. 
 :
:  GC-innhold (30%-52%).
GC-innhold (30%-52%).  a / U ≥ 3 i posisjoner 1-5.
a / U ≥ 3 i posisjoner 1-5.  Fravær av interne gjentakelser.
Fravær av interne gjentakelser.  A i posisjon 1.
A i posisjon 1.  A i posisjon 17.
A i posisjon 17.  U i posisjon 10.
U i posisjon 10.  Ikke G / C i posisjon 1.
Ikke G / C i posisjon 1.  Ikke G i posisjon 7.
Ikke G i posisjon 7. 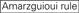 :
:  en asymmetri i stabiliteten til dupleksendene (målt som a / U-differensialet til de tre terminale baseparene i hver ende av duplekset).
en asymmetri i stabiliteten til dupleksendene (målt som a / U-differensialet til de tre terminale baseparene i hver ende av duplekset).  G Eller C i posisjon 19.
G Eller C i posisjon 19.  A eller U i posisjon 1.
A eller U i posisjon 1.  A eller U i posisjon 14.
A eller U i posisjon 14.  Ikke U i posisjon 19.
Ikke U i posisjon 19.  Ikke G i posisjon 1.
Ikke G i posisjon 1.
TRINN 2: Utvalg av siRNA-Sekvens Med Reduserte Off-Target-Effekter
for å unngå frøavhengige off-target-effekter, kan en tilnærming være å velge siRNA-styrestrengen hvis frøsekvens ikke er komplementær til noen sekvenser i 3 ‘ UTR av alle ikke-målrettede gener. Imidlertid er denne tilnærmingen vist seg å være umulig, fordi menneskelige sirnaer med den mest sjeldne syv – nt frøsekvensen fortsatt hadde frø-komplementariteter med flere ikke-målrettede mrnaer. Så vi har sett etter reglene som styrer siRNAs evne til å indusere frøavhengig off-target-effekt, og avslørte at effektiviteten av off-target-effekten er svært korrelert med den termodynamiske stabiliteten til duplekset dannet mellom frøområdet siRNA guide strand og dets mål mRNA (Ui-Tei et al., 2008). Smeltetemperaturen (Tm), en av de termodynamiske parametrene for dannelsen AV rna duplex, viste sterk positiv korrelasjon med induksjon av frøavhengige off-target-effekter. Dermed bør valg av siRNAs med lav Tm av frø – målet duplex minimere frøavhengig off-target silencing (Figur 1). Tm på 21,5°C kan tjene som referanse, som diskriminerer de nesten off-target-frie frøsekvensene fra de off-target-positive. Videre, siden off-target-effekten kan skyldes ikke bare av styrestrengen, men også av passasjerstrengen, er siRNAs hvis frømål Tm er tilstrekkelig lav for begge tråder gunstige.
TRINN 3: Eliminasjon Av Nesten Perfekt Matchet Off-Target Gener
Selv Når tm-verdien av frø-target duplexen er tilstrekkelig lav, kan målet gendeaktivering fortsatt finne sted hvis ikke-frø-regionen er fullstendig komplementær. Derfor, i det tredje trinnet, siRNAs som har nesten perfekte kamper til andre ikke-målrettede transkripsjoner ble eliminert (Figur 1).
siRNA Design Software
vi presenterte siRNA design software, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figur 2), som gir funksjonell, målspesifikk siRNA-designprogramvare i henhold til prosedyrene nevnt ovenfor (Naito et al., 2009). I standardparameteren kan siRNAs tilfredsstille Ui – tei-regelen velges. Når kandidatens funksjonelle sirnaer kan danne seed-target duplexes med tm-verdier under 21,5°C, og deres 19-nt-regioner som spenner over stillinger 2-20 av begge strengene, har minst to uoverensstemmelser med andre ikke-målrettede transkripsjoner, kan siDirect 2.0 designe minst en kvalifisert siRNA for >94% av menneskelige mRNA-sekvenser I RefSeq.

Figur 2. Skjermvisninger av siDirect 2.0 siRNA design software. (A) Toppside (http://siDirect2.RNAi.jp/). (B) Valgfrie parametere for siRNA design. (C) Resultat side. (D) Detaljert liste over off-target kandidater med nesten perfekte kamper. Justeringen mellom hver off-target transkripsjon og siRNA sekvensen visualiserer posisjonene til uoverensstemmelser.
Annen programvare for å velge funksjonelle sirnaer var åpen for publikum som vist I Tabell 1. I mange av dem, Ui-Tei regel (Ui-Tei et al.(2004), Reynolds rule (Reynolds et al., 2004), Amarzguioui-regelen (Amarzguioui og Prydz, 2004), Tuschl-regelen (Elbashir et al., 2002), og kombinasjonen av dem ble ofte og mye brukt. FOR å eliminere nesten perfekt matchet ikke-målet gener, BLAST søk ble brukt for homologi søk i flere programvare. Men SIDEN BLAST søk er ikke så nøyaktig for korte sekvenser som siRNAs, siDirect, wu-BLAST, Og Bowtie, som er svært nøyaktig homologi søkemotor for korte sekvenser blir ofte brukt. Blant dem kan siDirect 2.0 gi de mest nøyaktige resultatene. Videre noen av programvaren vurdere ekstra funksjoner, for eksempel mrna sekundær struktur (Ladunga, 2007; Lu Og Mathews, 2008), alternativ skjøting (Park et al., 2008), eller motivsekvensen der immunresponsen av ET RNA-virus kan oppstå (Gong et al., 2008). Disse funksjonene er ikke vurdert i siDirect 2.0. Så, for å ta hensyn til disse funksjonene, siRNAs vanligvis valgt av siDirect 2.0 og andre aktuelle programmer kan produsere optimalt resultat.
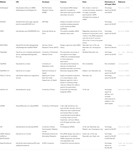
Tabell 1. Små forstyrrende RNA design programmer.
i tillegg er det praktisk talt viktig å bruke to eller flere sirnaer rettet mot forskjellige steder i et tiltenkt målgen, siden knockdown-effektene av et tiltenkt målgen er ment å være vanlige, men off-target-effektene vil sannsynligvis være forskjellige mellom sirnaer.
Konklusjon
effekten av hver siRNA er kjent for å variere mye avhengig av sekvensen i pattedyrceller, og bare en begrenset brøkdel av tilfeldig utformede sirna er funksjonell. Videre oppstår off-target silencing effekter når siRNA har delvis komplementaritet i frøområdet med utilsiktede gener. Her, basert På rnai-maskiner, beskrev vi den rasjonelle utformingen av funksjonelle, off-target-effektreduserte siRNAs, som forventes å slå ned et målgen-spesifikt.
Interessekonflikt
forfatterne erklærer at forskningen ble utført i fravær av kommersielle eller økonomiske forhold som kan tolkes som en potensiell interessekonflikt.
Takk
dette arbeidet ble delvis støttet av tilskudd Fra Departementet For Utdanning, Kultur, Sport, Vitenskap Og Teknologi I Japan (MEXT), Og Cell Innovation Project (MEXT), Og Kjerneforskningsprosjekt For Privat Universitet; matchende fondstilskudd Til Kumiko Ui-Tei.
Amarzguioui, M., Og Prydz, H. (2004). En algoritme for valg av funksjonelle siRNA-sekvenser. Biochem. Biophys. Res. Commun. 316, 1050–1058.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S., Og Khvorova, a. (2006). 3 ‘ UTR frø kamper, men ikke samlet identitet, er forbundet Med RNAi off-mål. Nat. Metoder 3, 199-204.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Castanotto, D., Og Rossi, J. j. (2009). Løftene og fallgruvene TIL RNA-interferensbasert terapi. Natur 457, 426-433.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Chalk, A. M. og Sonnhammer, E. L. L. (2008). siRNA spesifisitet søker innlemme mismatch toleranse data. Bioinformatikk 24, 1316-1317.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Cui, W., Ning, J., Naik, U. P., Og Duncan, M. K. (2004). OptiRNAi og rnai design tool. Comput. Metoder Programmer Biomed. 75, 67–73.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Ding, Y., Chan, C. Y. Og Lawrence, C. E. (2004). Sfold webserver for statistisk folding og rasjonell utforming av nukleinsyrer. Nukleinsyrer Res. 32, W135-W141.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K. og Tuschl, T. (2001). Duplekser av 21-nukleotid Rna medierer rna-interferens i dyrkede pattedyrceller. Natur 411, 494-498.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Elbashir, Wm, Harborth, J., Weber, K. og Tuschl, T. (2002). Analyse av genfunksjon i somatiske pattedyrceller ved bruk av små forstyrrende Rna. Metoder 26, 199-213.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Frank, F., Sonenberg, N., Og Nagar, B. (2010). Strukturell basis for 5 ‘ – nukleotid base-spesifikk anerkjennelse av guide RNA av human AGO2. Natur 465, 818-822.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Han er en av de eldste i Verden, Og er en av De eldste i Verden. (2008). siDRM: og effektivt og generelt anvendelig online siRNA designverktøy. Bioinformatikk 24, 2405-2406.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P. og Bartel, D. P. (2007). MicroRNA målretting spesifisitet i pattedyr: determinanter utover frø paring. Mol. Celle 27, 91-105.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R., Og Hannon, G. J. (2001). Argonaute 2 kobling mellom genetiske og biokjemiske analyser Av RNAi. Vitenskap 293, 1146-1150.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Henschel, A., Buchholz, F., Og Habermann, B. (2004). DEQOR: et nettbasert verktøy for design og kvalitetskontroll av siRNAs. Nukleinsyrer Res. 32, W113-W120.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Horn, T., Sandmann, T., Og Boutros, M. (2010). Design og evaluering av genom-brede biblioteker FOR RNA interferens skjermer. Genom Biol. 11, R61.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Jackson, A. L., Bartz, S. R., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., Og Linsley, P. S. (2003). Expression profilering avslører off-target genregulering Av RNAi. Nat. Bioteknologi. 21, 635–637.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L., Og Linsley, P. s. (2006). Utbredt siRNA «off-target» transkripsjon silencing mediert av frø region sekvens komplementaritet. RNA 12, 1179-1187.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Ketting, R. F. (2011). De mange ansiktene Til RNAi. Dev. Celle 15, 148-161.
Kryssref Fulltekst
Khvorova, A., Reynolds, A., Og Jayasena, S. D. (2003). Funksjonelle siRNAs og miRNAs viser strand bias. Celle 115, 209-216.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Ladunga, I. (2007). Mer komplett gendeaktivering med færre sirnaer: transparent optimalisert design og biofysisk signatur. Nukleinsyrer Res. 35, 433-440.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Leuschner, P. J., Ameres, S. L., Kueng, S., Og Martinez, J. (2006). Spaltning av siRNA passasjerstrengen under RISC-montering i humane celler. EMBO Rep. 7, 314-320.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Lewis, B. P., Burge, C. B. og Bartel, D. P. (2005). Konservert frøparing, ofte flankert av adenosiner, indikerer at tusenvis av menneskelige gener er mikrorna mål. Celle 120, 15-20.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. s., Og Johnson, J. M. (2005). Microarray analyse viser at noen microRNAs downregulate stort antall mål mrna. Natur 433, 769-773.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Lu, Z. J. Og Mathews, D. H. (2008). Effektiv siRNA utvalg ved hjelp av hybridisering termodynamikk. Nukleinsyre Res. 36, 640-647.
Kryssref Fulltekst
Ma, J.-B., Yuan, Yr, Meister, G., Pei, Y., Tuschl, T. og Patel, Dj (2005). Strukturelt grunnlag for 5 ‘ -end-spesifikk anerkjennelse av guide RNA Av a. fulgidus piwi protein. Natur 434, 666-670.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Martinez, J., Patkaniowska, a., Urlaub, H., Luhrmann, R., Og Tuschl, T. (2002). Single-strandet antisense siRNAs guide målet RNA spaltning I RNAi. Celle 110, 563-574.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Matranga, C., Tomari, Y., Shin, C., Bartel, Dp, Og Zamore, Pd (2005). Passasjer-strand spaltning letter montering av siRNA I Ago2-holdige rnai enzymkomplekser. Celle 123, 607-620.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Naito, Y., Yoshimura, J., Morishita, S., Og Ui-Tei, K. (2009). siDirect 2.0: oppdatert programvare for å designe funksjonell siRNA med redusert frøavhengig off-target effekt. BMC Bioinformatikk 10, 392.
Kryssref Fulltekst
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M., Og Kim, Y. J. (2008). AsiDesigner: exon-basert siRNA design server vurderer alternativ spleising. Nukleinsyre Res. 36, W97-W103.
Kryssref Fulltekst
Rand, T. A., Petersen, S., Du, F., Og Wang, X. (2005). Argonaute2 spalter anti-guide strand av siRNA UNDER RISC aktivering. Celle 123, 621-629.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, Ws, Og Khvorova, a. (2004). Rasjonell siRNA-design for rna-interferens. Nat. Bioteknologi. 22, 326–330.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N., Og Zamore, P. D. (2003). Asymmetri i samlingen Av rnai enzymkomplekset. Celle 115, 199-208.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Shah, Jk, Garner, Hr, White, Ma, Shames, Dsog Minna, Jd (2007). sirna information resource, et web – basert verktøy for siRNA sekvens design og analyse og en åpen tilgang siRNA database. BMC Bioinformatikk 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Full Text
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A. og Saigo, K. (2008). Termodynamisk stabilitet og watoson-Crick base paring i frø duplex er viktige faktorer som bestemmer effektiviteten av sRNA-basert off-target effekt. Nukleinsyrer Res. 36, 7100-7109.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R. og Saigo, K. (2004). Retningslinjer for valg av svært effektive siRNA sekvenser for pattedyr OG chick RNA interferens. Nukleinsyrer Res. 32, 936-948.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Vert, J.-P., Foveau, N., Lajaunie, C., Og Vandenbrouck, Y. (2006). En nøyaktig og tolkbar modell for sirna effekt prediksjon. BMC Bioinformatikk 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Full Text
Yuan, B., Latek, R., Rossbach, M., Tuschl, T. og Lewitter, F. (2004). siRAN selection server: en automatisert siRNA oligonukleotid prediksjon server. Nukleinsyrer Res. 32, W130-W134.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst
Yuan, Y.-R., Pei, Y., Ma, Jb, Kuryavyi, V., Zhandina, M., Meister, G., Chen, Hy, Dauter, Z., Tuschl, T. og Patel, Dj (2005). Krystallstruktur Av A. aeolicus argonaute, en stedsspesifikk DNA-guidet endoribonuklease, gir innsikt I RISC-mediert mRNA spaltning. Mol. Celle 19, 405-419.
Pubmed Abstrakt / Pubmed Fulltekst / CrossRef Fulltekst