- wprowadzenie
- Zoptymalizowana konstrukcja Sirna
- Krok 1: Wybór funkcjonalnej sekwencji siRNA
- Krok 2: Wybór sekwencji siRNA ze zmniejszonymi efektami poza-docelowymi
- Krok 3: Eliminacja prawie perfekcyjnie dopasowanych genów poza celem
- oprogramowanie siRNA Design Software
- wniosek
- Oświadczenie o konflikcie interesów
- podziękowania
wprowadzenie
interferencja RNA (RNAi) jest szeroko stosowaną techniką, dzięki której mały interferujący RNA (siRNA) obniża specyficzny gen docelowy o doskonałej komplementarnej sekwencji i obiecuje zastosowanie terapeutyczne w chorobach ludzkich (Castanotto and Rossi, 2009; Ketting, 2011). Chociaż człowiek ma ponad 20 000 genów, pożądane jest użycie siRNA, który jest wysoce funkcjonalny i nie ma wpływu na żadne geny inne niż jego specyficzny cel. W tym artykule omówimy zoptymalizowaną metodę projektowania siRNA w oparciu o mechanizm RNAi. Ponadto przedstawiamy ogólnodostępne strony internetowe do wyboru sekwencji siRNA.
Zoptymalizowana konstrukcja Sirna
dupleksów 21-nukleotydowego (nt) RNA z 2 nt 3′ nawisami (siRNA) jest zwykle używana do eksperymentów RNAi. Po dostarczeniu do komórek, sirna są włączane do RNA-induced silencing complex (RISC) jako dwuniciowy RNA. RISC to kompleks efektorowy zawierający białko Argonaute (Ago) o aktywności krajalnicy (Hammond et al., 2001; Martinez et al., 2002). Nić przewodnika siRNA zawierająca termodynamicznie mniej stabilny 5 ’ – koniec jest preferencyjnie zachowywana przez RISC(Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). Nici pasażerskie większości dwuniciowych sirna załadowanych na RISC są rozszczepiane przez białko Ago2 i rozkładane (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). Zatrzymane pary nici prowadzącej celują w mRNA z doskonale komplementarną sekwencją i tłumią ją przez rozszczepianie przez białko Ago2 w pozycji nukleotydowej 10 nici prowadzącej siRNA (Elbashir i in., 2001; Hammond et al., 2001; Martinez et al., 2002). Jednak zgromadzone dowody z eksperymentów obejmujących cały genom wskazują, że duża liczba mRNA z częściowymi komplementarnościami do nici przewodnika jest również zmniejszona (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Zjawisko to określa się jako efekt uboczny zależny od nasion i korzystnie obserwuje się w mRNA 3′ UTRs. Mechanizm rozpoznawania celu tego efektu poza celem jest podobny do mechanizmu wyciszania genów za pośrednictwem miRNA (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Transkrypty z sekwencjami komplementarnymi do regionu nasiennego położonego 2-8 od końca 5′ są głównie zredukowane. Wiadomo, że region nasion znajduje się na powierzchni Ago w formie quasi-spiralnej, aby służyć jako miejsce wejścia lub zarodkowania dla małych RNA w RISCs(Ma et al., 2005; Yuan et al., 2005). Tak więc, Region nasion najpierw identyfikuje docelowe mRNA, a następnie tworzy idealne parowanie bazy z zamierzonym docelowym mRNA i indukuje RNAi przez Ago2.
opierając się na mechanizmie RNAi, siRNA swoiste dla danego genu docelowego uważa się za selekcyjne zgodnie z następującymi trzema etapami.
Krok 1: Wybór funkcjonalnej sekwencji siRNA
okazuje się, że skuteczność strącania sirna jest w dużym stopniu zależna od ich sekwencji. Opowiadaliśmy się za empirycznie opartą regułą, która przepisywała cechy wysoce funkcjonalnych sirna (Ui-Tei et al., 2004), taka reguła nazywana jest regułą Ui-Tei (Rys. 1). SiRNA wybrane przez regułę Ui-Tei spełniają jednocześnie następujące cztery warunki: (1) A lub U w pozycji 1 od 5′ końca nici prowadzącej siRNA, (2) g lub C w pozycji 19, (3) bogactwo AU (AU ≥4) w pozycjach 1-7 oraz (4) Brak długiego odcinka GC ≥10. Z wyjątkiem (4), nasza zasada wskazywała, że funkcjonalny siRNA ma asymetryczną stabilność w zaciskach 5′ I 3′. Nasza eksperymentalna Walidacja z użyciem testu reportera lucyferazy wykazała, że 98% sirna spełniających powyższe warunki zmniejszyło ekspresję reportera lucyferazy poniżej 33% (Ui-Tei i wsp., 2004). Inne grupy wykazały również zasady wysoce funkcjonalnych sirna określane jako reguła Reynoldsa (Reynolds et al., 2004) i reguła Amarzguioui (amarzguioui i Prydz, 2004), jak podsumowano na rysunku 1. Zasady te wyraźnie pokazały również, że funkcjonalne sirna są asymetryczne: nić RNA z niestabilnym terminalem 5′ była skuteczna jako nić prowadząca. Ponadto, powszechne w tych zasadach, 5 ’ terminus funkcjonalnej nici przewodnika siRNA było lepsze być A lub U. okazało się później, że wyniki odzwierciedlają cechy strukturalne ludzkiego Ago2 (Frank et al., 2010). Struktura krystaliczna domeny Środkowej (Środkowej) z ludzkich eksperymentów miareczkowania AGO2 i NMR wykazała, że monofosforany nukleotydów, AMP i UMP, wiążą się z nawet 30-krotnie większym powinowactwem niż CMP lub GMP, dostarczając strukturalnych dowodów na specyficzne dla nukleotydów interakcje w środkowej domenie białek eukariotycznych AGO.
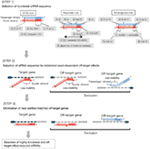
Rysunek 1. Schematyczna reprezentacja do wyboru funkcjonalnych i poza-docelowych sirna zmniejszonych efektów. Wybór wysoce funkcjonalnego siRNA według reguł Ui-Tei, Reynoldsa, Amarzguioui lub ich kombinacji (Krok 1). Selekcja sirna o niskiej stabilności w dupleksach nasiennych (etap 2). Eliminacja sirna z prawie perfekcyjnie dopasowanymi sekwencjami do genów innych niż docelowe (etap 3). W każdej z reguł pozycja nukleotydu wskazuje liczbę nukleotydów liczoną od 5 ’ końca nici prowadzącej.  :
:  A / U na pozycji 1.
A / U na pozycji 1.  G / C na pozycji 19.
G / C na pozycji 19.  4 do 7 a / Us na pozycjach 1-7.
4 do 7 a / Us na pozycjach 1-7. 
 :
:  zawartość GC (30% -52%).
zawartość GC (30% -52%).  A/U ≥ 3 na pozycjach 1-5.
A/U ≥ 3 na pozycjach 1-5.  brak powtórzeń wewnętrznych.
brak powtórzeń wewnętrznych.  A na pozycji 1.
A na pozycji 1.  a na pozycji 17.
a na pozycji 17.  U na pozycji 10.
U na pozycji 10.  Nie G / C na pozycji 1.
Nie G / C na pozycji 1.  Nie G na pozycji 7.
Nie G na pozycji 7. 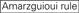 :
:  asymetria stabilności końców dupleksu (mierzona jako różnica A/U trzech końcowych par bazowych na obu końcach dupleksu).
asymetria stabilności końców dupleksu (mierzona jako różnica A/U trzech końcowych par bazowych na obu końcach dupleksu).  G lub C na pozycji 19.
G lub C na pozycji 19.  A lub U na pozycji 1.
A lub U na pozycji 1.  A lub U na pozycji 14.
A lub U na pozycji 14.  Nie U na pozycji 19.
Nie U na pozycji 19.  Nie G na pozycji 1.
Nie G na pozycji 1.
Krok 2: Wybór sekwencji siRNA ze zmniejszonymi efektami poza-docelowymi
aby uniknąć efektów poza-docelowych zależnych od nasion, jednym z podejść może być wybranie nici prowadzącej siRNA, której Sekwencja nasion nie jest komplementarna do żadnej sekwencji w 3′ UTR wszystkich genów niecelowanych. Jednak to podejście okazało się niemożliwe, ponieważ ludzkie sirna z najbardziej rzadką sekwencją nasion o siedmiowartościowej sekwencji nasiennej nadal miały komplementarność nasienną z kilkoma niecelowanymi mRNA. Tak więc, szukaliśmy zasad regulujących zdolność sirna do indukowania zależnego od nasion efektu poza celem i ujawniliśmy, że skuteczność efektu poza celem jest wysoce skorelowana ze stabilnością termodynamiczną dupleksu utworzonego między regionem nasion nici prowadzącej siRNA a jej docelowym mRNA (Ui-Tei et al., 2008). Temperatura topnienia (Tm), jeden z parametrów termodynamicznych do tworzenia dupleksu RNA, wykazywał silną dodatnią korelację z indukcją zależnych od nasion efektów poza celem. Tak więc wybór sirna o niskim Tm dupleksu docelowego nasion powinien zminimalizować uciszanie poza celem zależne od nasion (Rys. 1). TM wynoszący 21,5°C może służyć jako punkt odniesienia, który odróżnia sekwencje nasion prawie bez celu od sekwencji nasion poza celem dodatnich. Ponadto, ponieważ efekt poza celem może być spowodowany nie tylko przez nić prowadzącą, ale także przez nić pasażerską, siRNA, których docelowy TM nasion jest wystarczająco niski dla obu nici, są korzystne.
Krok 3: Eliminacja prawie perfekcyjnie dopasowanych genów poza celem
nawet gdy wartość TM dupleksu docelowego nasion jest wystarczająco niska, wyciszanie docelowego genu może nadal mieć miejsce, jeśli region Nie-nasienny jest całkowicie komplementarny. Dlatego w trzecim etapie wyeliminowano sirna, które mają niemal idealne dopasowanie do wszelkich innych transkryptów, które nie są ukierunkowane(rycina 1).
oprogramowanie siRNA Design Software
zaprezentowaliśmy oprogramowanie siRNA design software, siDirect 2.0 (http://siDirect2.RNAi.jp/ ; Rysunek 2), który zapewnia funkcjonalne, specyficzne dla celu oprogramowanie do projektowania siRNA zgodnie z procedurami wymienionymi powyżej(Naito et al., 2009). W parametrze domyślnym można wybrać sirna spełniające regułę Ui-Tei. Gdy kandydujące funkcjonalne sirna mogą tworzyć dupleksy seed-target o wartościach TM poniżej 21,5°C, a ich 19-nt regiony obejmujące pozycje 2-20 obu nici mają co najmniej dwa niedopasowania do innych niekierowanych transkryptów, siDirect 2.0 może zaprojektować co najmniej jeden kwalifikowany siRNA dla > 94% ludzkich sekwencji mRNA w RefSeq.

Rysunek 2. Widok ekranu oprogramowania sidirect 2.0 siRNA design. A) Strona główna (http://siDirect2.RNAi.jp/). B) parametry opcjonalne dla konstrukcji siRNA. (C) Strona wyników. (D) szczegółowa lista kandydatów spoza grupy docelowej z dopasowaniem zbliżonym do idealnego. Wyrównanie pomiędzy każdym transkryptem poza celem a sekwencją siRNA wizualizuje pozycje niedopasowań.
inne oprogramowanie do wyboru funkcjonalnych sirna było publicznie dostępne, jak pokazano w tabeli 1. W wielu z nich zasada Ui-Tei (Ui-Tei et al., 2004), reguła Reynoldsa (Reynolds et al., 2004), reguła Amarzguioui (amarzguioui i Prydz, 2004), reguła Tuschla (Elbashir et al., 2002), a ich połączenie było często i szeroko stosowane. Aby wyeliminować prawie idealnie dopasowane geny inne niż docelowe, BLAST search został użyty do wyszukiwania homologii w kilku programach. Jednakże, ponieważ wyszukiwanie BLAST nie jest tak dokładne dla krótkich sekwencji, takich jak siRNAs, siDirect, WU-BLAST i Bowtie, które są bardzo dokładne wyszukiwarka homologii dla krótkich sekwencji są często używane. Wśród nich, sidirect 2.0 może przynieść najbardziej dokładne wyniki. Ponadto, niektóre z oprogramowania rozważyć dodatkowe funkcje, takie jak wtórna struktura mRNA (Ladunga, 2007; Lu i Mathews, 2008), alternatywne splicing (Park et al., 2008), lub sekwencji motywów, za pomocą których może wystąpić odpowiedź immunologiczna wirusa RNA (Gong et al., 2008). Funkcje te nie są brane pod uwagę w siDirect 2.0. Tak więc, aby wziąć pod uwagę te cechy, sirna powszechnie wybierane przez siDirect 2.0 i inne odpowiednie programy mogą dać optymalny wynik.
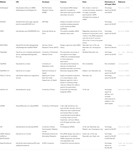
Tabela 1. Małe interferujące programy do projektowania RNA.
ponadto, praktycznie ważne jest, aby używać dwóch lub więcej sirna ukierunkowanych na różne miejsca w zamierzonym Genie docelowym, ponieważ efekty knockdown zamierzonego genu docelowego są prawdopodobnie powszechne, ale efekty poza celem są prawdopodobnie różne między sirna.
wniosek
wiadomo, że skuteczność każdego siRNA jest bardzo różna w zależności od jego sekwencji w komórkach ssaków i tylko ograniczona część losowo zaprojektowanych sirna jest funkcjonalna. Co więcej, efekty wyciszania poza celem pojawiają się, gdy siRNA ma częściową komplementarność w regionie nasion z niezamierzonymi genami. Tutaj, na podstawie maszynerii RNAi, opisaliśmy racjonalną konstrukcję funkcjonalnych, pozbawionych efektu docelowego sirna, które mają powalić docelowy Gen.
Oświadczenie o konflikcie interesów
autorzy oświadczają, że badanie zostało przeprowadzone przy braku jakichkolwiek relacji handlowych lub finansowych, które mogłyby być interpretowane jako potencjalny konflikt interesów.
podziękowania
ta praca była częściowo wspierana przez granty z Ministerstwa Edukacji, Kultury, Sportu, Nauki i technologii Japonii (MEXT), i Cell Innovation Project (MEXT), i główny projekt badawczy dla prywatnego uniwersytetu; subwencja funduszu dopasowującego do Kumiko Ui-Tei.
Amarzguioui, M., and Prydz, H. (2004). Algorytm doboru funkcjonalnych sekwencji siRNA. Biochem. Biophys. Res.Commun. 316, 1050–1058.
PubMed Streszczenie / PubMed Pełny Tekst / CrossRef Pełny Tekst
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpiłow, J., Marshall, W. S., and Khvorova, A. (2006). 3 ’ UTR Seed mecze, ale nie ogólna tożsamość, są związane z RNAi off-targets. Nat. Metody 3, 199-204.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Castanotto, D., and Rossi, J. J. (2009). Obietnice i pułapki terapii opartych na interferencji RNA. Nature 457, 426-433.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
kreda, A. M., and Sonnhammer, E. L. L. (2008). wyszukiwanie specyficzności siRNA obejmujące dane tolerancji niedopasowania. Bioinformatyka 24, 1316-1317.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Cui, W., Ning, J., Naik, U. P., and Duncan, M. K. (2004). OptiRNAi i RNAi design tool. Comput. Metody Programy Biomed. 75, 67–73.
PubMed Streszczenie / PubMed Pełny Tekst / CrossRef Pełny Tekst
Ding, Y., Chan, C. Y., and Lawrence, C. E. (2004). Sfold web serwer do statystycznego składania i racjonalnego projektowania kwasów nukleinowych. Kwasy Nukleinowe Res. 32, W135–W141.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K., and Tuschl, T. (2001). Dupleksy 21-nukleotydowych RNA pośredniczą w interferencji RNA w hodowanych komórkach ssaków. Nature 411, 494-498.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Elbashir, W. M., Harborth, J., Weber, K., and Tuschl, T. (2002). Analiza funkcji genów w somatycznych komórkach ssaków z wykorzystaniem małych interferujących RNA. Metody 26, 199-213.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Frank, F., Sonenberg, N., And Nagar, B. (2010). Strukturalna podstawa dla 5 ’ -nukleotydowego rozpoznawania RNA przewodnika przez ludzki AGO2. Nature 465, 818-822.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S., and Li, Tongbin. (2008). siDRM: oraz skuteczne i powszechnie stosowane narzędzie do projektowania siRNA online. Bioinformatyka 24, 2405-2406.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P., and Bartel, D. P. (2007). MikroRNA ukierunkowana na specyficzność u ssaków: determinanty wykraczające poza parowanie nasion. Mol. Kom. 27, 91-105
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R., and Hannon, G. J. (2001). Argonaute 2 związek między genetycznych i biochemicznych analiz RNAi. Nauka 293, 1146-1150.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Henschel, A., Buchholz, F., And Habermann, B. (2004). DEQOR: internetowe narzędzie do projektowania i kontroli jakości sirna. Kwasy Nukleinowe Res. 32, W113–W120.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Horn, T., Sandmann, T., and Boutros, M. (2010). Projektowanie i ocena bibliotek genomu dla ekranów interferencyjnych RNA. Genom Biol. 11, R61.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Jackson, A. L., Bartz, S. R., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., and Linsley, P. S. (2003). Profilowanie ekspresji ujawnia regulację genów poza celem przez RNAi. Nat. Biotechnol. 21, 635–637.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L., and Linsley, P. S. (2006). Powszechne wyciszanie transkrypcji siRNA „poza celem” za pośrednictwem komplementarności sekwencji regionu nasiennego. 1179-1187
PubMed Streszczenie / PubMed Pełny Tekst / CrossRef Pełny Tekst
Ketting, R. F. (2011). Wiele twarzy RNAi. Dev. Cela 15, 148-161.
CrossRef Pełny tekst
Khvorova, A., Reynolds, A., and Jayasena, S. D. (2003). Funkcjonalne sirna i mirna wykazują odchylenie nici. Cela 115, 209-216.
PubMed Streszczenie / PubMed Pełny Tekst / CrossRef Pełny Tekst
Ladunga, I. (2007). Pełniejsze wyciszanie genów przez mniejszą liczbę sirna: transparentna zoptymalizowana konstrukcja i sygnatura biofizyczna. Nucleic Acids Res.35, 433-440.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Leuschner, P. J., Ameres, S. L., Kueng, S., and Martinez, J. (2006). Rozszczepienie nici pasażera siRNA podczas montażu RISC w komórkach ludzkich. EMBO Rep. 7, 314-320.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
(2005) Zachowane parowanie nasion, często otoczone przez adenozyny, wskazuje, że tysiące ludzkich genów są celami mikroRNA. Cela 120, 15-20.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S., and Johnson, J. M. (2005). Analiza mikromacierzy pokazuje, że niektóre mikroRNA zmniejszają dużą liczbę docelowych mRNA. Nature 433, 769-773.
PubMed Streszczenie / PubMed Pełny Tekst / CrossRef Pełny Tekst
Lu, Z. J., and Mathews, D. H. (2008). Efektywna selekcja siRNA z wykorzystaniem termodynamiki hybrydyzacji. Nucleic Acid Res. 36, 640-647.
CrossRef Pełny tekst
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T., and Patel, D. J. (2005). Strukturalne podstawy dla 5 ’ – endowego specyficznego rozpoznawania RNA przewodnika przez białko A. fulgidus piwi. Nature 434, 666-670.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R., and Tuschl, T. (2002). Jednoniciowe antysensowne sirna kierują rozszczepianiem docelowego RNA w RNAi. Cela 110, 563-574.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., and Zamore, P. D. (2005). Rozszczep nici pasażera ułatwia montaż siRNA w kompleksy enzymatyczne RNAi zawierające Ago2. Kom. 123, 607-620.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Naito, Y., Yoshimura, J., Morishita, S., and Ui-Tei, K. (2009). sidirect 2.0: zaktualizowane oprogramowanie do projektowania funkcjonalnego siRNA ze zmniejszonym efektem poza-docelowym zależnym od nasion. BMC Bioinformatics 10, 392.
CrossRef Pełny tekst
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M., and Kim, Y. J. (2008). AsiDesigner: serwer siRNA oparty na exonie, rozważający alternatywne splicing. Kwas Nukleinowy Res. 36, W97-W103.
CrossRef Pełny tekst
Rand, T. A., Petersen, S., Du, F., and Wang, X. (2005). Argonaute2 rozszczepia nić anty-przewodnika siRNA podczas aktywacji RISC. Kom. 123, 621-629.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S., and Khvorova, A. (2004). Racjonalna konstrukcja siRNA dla interferencji RNA. Nat. Biotechnol. 22, 326–330.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N., and Zamore, P. D. (2003). Asymetria w montażu kompleksu enzymatycznego RNAi. Cela 115, 199-208.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Shah, J. K., Garner, H. R., White, M. A., Shames, D. S., and Minna, J. D. (2007). sirna information resource, internetowe narzędzie do projektowania i analizy sekwencji siRNA oraz otwarta baza danych siRNA. BMC Bioinformatyka 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Pełny tekst
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A., and Saigo, K. (2008). Stabilność termodynamiczna i parowanie bazy Watoson-Crick w dupleksie nasion są głównymi wyznacznikami skuteczności efektu poza-docelowego opartego na sRNA. Nucleic Acids Res. 36, 7100-7109.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R., and Saigo, K. (2004). Wytyczne dotyczące wyboru wysoce skutecznych sekwencji siRNA dla interferencji RNA ssaków i kurcząt. Nucleic Acids Res. 32, 936-948.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Vert, J.-P., Foveau, N., Lajaunie, C., and Vandenbrouck, Y. (2006). Dokładny i interpretowalny model przewidywania skuteczności siRNA. BMC Bioinformatics 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Pełny tekst
Yuan, B., Latek, R., Rossbach, M., Tuschl, T., and Lewitter, F. (2004). siRAN selection server: zautomatyzowany serwer przewidywania oligonukleotydów siRNA. Kwasy Nukleinowe Res. 32, W130–W134.
PubMed Streszczenie / PubMed Pełny tekst / CrossRef Pełny tekst
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T., and Patel, D. J. (2005). Struktura krystaliczna A. aeolicus argonaute, endoribonukleazy kierowanej przez DNA, zapewnia wgląd w rozszczepianie mRNA za pośrednictwem RISC. Mol. Cela 19, 405-419.
PubMed Abstract / PubMed Full Text / CrossRef Full Text