- Introdução
- Design optimizado de siRNA
- STEP 1: Selection of Functional siRNA Sequence
- Passo 2: Selecção da sequência siRNA com efeitos fora do alvo reduzidos
- Passo 3: Eliminação de Genes Off-Target quase-perfeitos combinados
- siRNA Design Software
- conclusão
- Declaração de conflito de interesses
- Agradecimentos
Introdução
RNA de interferência (RNAi) é uma técnica amplamente utilizada pelos quais a pequena interferência de RNA (siRNA) downregulates um determinado gene alvo com perfeita complementares sequência, e prometeu usar na aplicação terapêutica de doenças humanas (Castanotto e Rossi, 2009; Ketting, 2011). Embora o ser humano tenha mais de 20.000 genes, é desejável usar siRNA que é altamente funcional e não tem efeitos em quaisquer genes além do seu alvo específico. Neste artigo, revisamos um método otimizado para projetar siRNA baseado no mecanismo da RNAi. Além disso, introduzimos os sites abertos ao público para selecionar sequências siRNA.
Design optimizado de siRNA
Duplexs of 21-nucleotide (nt) RNA with 2 nt 3′ overhangs (siRNA) is usually used for RNAi experiments. Após a entrega nas células, siRNAs são incorporadas ao complexo silenciador induzido pelo ARN (RISC) como um RNA de cadeia dupla. RISC é o complexo efetor contendo proteína Argonaute (Ago) com atividade cortadora (Hammond et al., 2001; Martinez et al., 2002). A cadeia guia siRNA que contém a extremidade termodinamicamente menos estável de 5’é preferencialmente retida por RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). As cadeias de passageiros da maior parte das siRNAs de cadeia dupla carregadas no RISC são clivadas pela proteína Ago2 e degradadas (Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). A cadeia guia retida tem pares de ARNm com sequência perfeitamente complementar, e reprime-a pela clivagem da proteína Ago2 na posição nucleótida 10 da cadeia guia siRNA (Elbashir et al., 2001; Hammond et al., 2001; Martinez et al., 2002). No entanto, uma evidência acumulada de experimentos em todo o genoma indica que um grande número de mRNAs com complementaridades parciais à linha guia também são reduzidas (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Este fenómeno é referido como efeito off-target dependente das sementes e, de preferência, observado no mRNA 3′ UTRs. O mecanismo de reconhecimento alvo deste efeito fora do alvo é conhecido por ser semelhante ao do silenciamento genético mediado por miRNA (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). As transcrições com sequências complementares à região de sementes posicionadas 2-8 a partir do terminal 5′ são principalmente reduzidas. A região de sementes é conhecida por estar situada na superfície de Ago em uma forma quase-helicoidal para servir como local de entrada ou nucleação para pequenas RNAs nos RISCs (Ma et al., 2005; Yuan et al., 2005). Assim, a região de sementes primeiro identifica o alvo mRNAs, e, posteriormente, formam um par perfeito de base com o alvo pretendido mRNA e induzem RNAi por Ago2.
com base no mecanismo de RNAi, um siRNA específico do gene alvo é considerado seleccionável de acordo com os três passos seguintes.
STEP 1: Selection of Functional siRNA Sequence
The knockdown eficiências of siRNAs are revealed to be highly dependent on their sequences. Nós defendemos a regra empiricamente baseada que prescrevia as características de siRNAs altamente funcionais (Ui-Tei et al., 2004), tal regra é chamada de regra Ui-Tei (Figura 1). O siRNA seleccionado pela regra Ui-Tei satisfaz simultaneamente as quatro condições seguintes:: (1) A ou U na posição 1 a partir de 5′ terminus do cabo de siRNA guide strand, (2) g ou C na posição 19, (3) AU richness (AU ≥4) nas posições 1-7 e (4) no long GC stretch ≥10. Com exceção de (4), nossa regra indicava que o siRNA funcional tem estabilidade assimétrica em terminais de 5′ e 3′. A nossa validação experimental com o ensaio de luciferase reporter mostrou que 98% das siRNAs que satisfazem as condições acima reduziram a expressão de luciferase reporter a menos de 33% (Ui-Tei et al., 2004). Outros grupos também demonstraram as regras de siRNAs altamente funcionais referidas como regra de Reynolds (Reynolds et al., 2004) e a regra Amarzguioui (Amarzguioui e Prydz, 2004) como resumida na Figura 1. Estas regras também mostraram claramente que as siRNAs funcionais são assimétricas: uma cadeia de RNA com um terminal instável de 5′ era eficaz como uma cadeia guia. Além disso, comum a estas regras, 5′ terminus do funcional de siRNA guia vertente era preferível ser Um ou U. foi revelado mais tarde que o resultado reflete as características estruturais de humanos Ago2 (Frank et al., 2010). A estrutura cristalina de um domínio médio (médio) de experimentos de titulação de AGO2 e NMR humanos mostrou que os monofosfatos de nucleótidos, AMP e UMP, se ligam com afinidade 30 vezes maior do que CMP ou GMP, fornecendo evidências estruturais para interações específicas de nucleótidos no domínio médio das proteínas eucarióticas AGO.
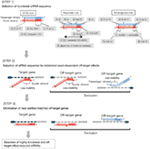
Figura 1. Representação esquemática para seleccionar siRNAs funcionais e fora do alvo. Seleção de siRNA altamente funcional por Ui-Tei, Reynolds, Amarzguioui regras, ou a combinação deles (Passo 1). Selecção de siRNAs com baixa estabilidade nos duplexes do alvo de sementes (Passo 2). Eliminação de siRNAs com sequências quase perfeitas para genes não-alvo (Passo 3). Em cada regra, a posição do nucleótido indica o número de nucleótidos contados a partir do Terminal 5′ da cadeia guia. 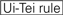 :
:  a / U na posição 1.
a / U na posição 1.  G / C na posição 19.
G / C na posição 19.  4 a 7 a / Us nas posições 1-7.
4 a 7 a / Us nas posições 1-7. 
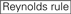 :
:  teor de GC (30% -52%).
teor de GC (30% -52%).  a / U ≥ 3 nas posições 1-5.
a / U ≥ 3 nas posições 1-5.  ausência de repetições internas.
ausência de repetições internas.  A na posição 1.
A na posição 1.  A na posição 17.
A na posição 17.  U na posição 10.
U na posição 10.  Não G/C na posição 1.
Não G/C na posição 1.  Não G Na posição 7.
Não G Na posição 7.  :
:  uma assimetria na estabilidade das extremidades duplex (medida como o diferencial A/U dos três basepairs terminais em cada extremidade do duplex).
uma assimetria na estabilidade das extremidades duplex (medida como o diferencial A/U dos três basepairs terminais em cada extremidade do duplex).  G Ou C na posição 19.
G Ou C na posição 19.  A ou U na posição 1.
A ou U na posição 1.  A ou U na posição 14.
A ou U na posição 14.  não U na posição 19.
não U na posição 19.  Não G Na posição 1.
Não G Na posição 1.
Passo 2: Selecção da sequência siRNA com efeitos fora do alvo reduzidos
para evitar efeitos fora do alvo dependentes de sementes, uma abordagem pode ser a selecção da cadeia guia siRNA cuja sequência de sementes não é complementar a quaisquer sequências nos 3′ UTR de todos os genes não visados. No entanto, esta abordagem provou ser impossível, porque as siRNAs humanas com a sequência de sementes de sete nt mais rara ainda tinham complementaridades de sementes com vários mRNAs não visados. Assim, temos procurado as regras que regem a capacidade de siRNAs para induzir o efeito off-target dependente de sementes, e revelou que a eficiência do efeito off-target é altamente correlacionada com a estabilidade termodinâmica do duplex formado entre a região de sementes de siRNA guide strand e seu alvo mRNA (Ui-Tei et al., 2008). A temperatura de fusão (Tm), um dos parâmetros termodinâmicos para a formação de ARN duplex, mostrou forte correlação positiva com a indução de efeitos dependentes de sementes fora do alvo. Assim, a seleção das siRNAs com Tm baixo do duplex do alvo-semente deve minimizar o silenciamento fora do alvo dependente da semente (Figura 1). O Tm de 21,5 ° C pode servir de referência, o que discrimina as sequências de sementes quase fora do alvo das sequências de sementes fora do alvo positivas. Além disso, uma vez que o efeito off-target pode ser causado não só pela linha guia, mas também pela linha de passageiros, siRNAs cujo TM-alvo de sementes é suficientemente baixo para ambas as cadeias são favoráveis.
Passo 3: Eliminação de Genes Off-Target quase-perfeitos combinados
mesmo quando o valor Tm do duplex do alvo-semente é suficientemente baixo, o silenciamento do gene-alvo ainda pode ocorrer se a região não-semente for completamente complementar. Portanto, na terceira etapa, as siRNAs que têm correspondências quase perfeitas a quaisquer outras transcrições não direcionadas foram eliminadas (Figura 1).
siRNA Design Software
we presented siRNA design software, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figura 2), que fornece software de design siRNA funcional e específico de destino de acordo com os procedimentos acima mencionados (Naito et al., 2009). In default parameter, siRNAs satisfying Ui-Tei rule can be selected. Quando o candidato funcional siRNAs poderia formulário de sementes-alvo duplex com Tm valores abaixo de 21,5°C, e os seus 19-nt regiões de abrangência de posições 2-20 de ambas as vertentes têm, pelo menos, dois inadequação para qualquer outro não-alvo transcrições, siDirect 2.0 pode criar pelo menos um qualificado siRNA para >94% dos humanos seqüências de mRNA em RefSeq.

Figura 2. Screen views of siDirect 2.0 siRNA design software. (A) Top page (http://siDirect2.RNAi.jp/). B) parâmetros facultativos para a concepção do siRNA. (C) página do resultado. D) lista pormenorizada dos candidatos não visados com resultados quase perfeitos. O alinhamento entre cada transcrição fora do alvo e a sequência siRNA visualiza as posições de desfasamentos.
outros softwares para selecionar siRNAs funcionais foram abertos ao público como mostrado na Tabela 1. Em muitos deles, a regra Ui-Tei (Ui-Tei et al., 2004), Reynolds rule (Reynolds et al., 2004), Amarzguioui rule (Amarzguioui and Prydz, 2004), Tuschl rule (Elbashir et al., 2002), e a sua combinação foi frequente e amplamente utilizada. Para eliminar os genes não-alvo quase perfeitos, a busca por explosão foi usada para pesquisa de homologia em vários softwares. No entanto, uma vez que a busca de explosão não é tão precisa para sequências curtas como siRNAs, siDirect, WU-BLAST, e Bowtie, que são altamente precisos motor de busca de homologia para sequências curtas são frequentemente usados. Entre eles, siDirect 2.0 pode trazer os resultados mais precisos. Além disso, alguns dos softwares consideram as características adicionais, como a estrutura secundária mRNA (Ladunga, 2007; Lu e Mathews, 2008), splicing alternativo (Park et al., 2008), ou a sequência motif pela qual a resposta imunitária por um vírus RNA pode ocorrer (Gong et al., 2008). Estas características não são consideradas no siDirect 2.0. Assim, para levar em conta essas características, siRNAs comumente selecionados pela siDirect 2.0 e os outros programas de software apropriados podem produzir um resultado ótimo.
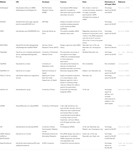
Quadro 1. Pequenos programas de design de RNA interferentes.
além disso, é praticamente consideração importante a utilização de dois ou mais siRNAs segmentação de diferentes sites em um alvo gene, desde a queda de efeitos de um alvo o gene é suposto ser o comum, mas o off-alvo, os efeitos podem ser diferentes entre siRNAs.
conclusão
sabe-se que a eficácia de cada siRNA varia muito dependendo da sua sequência em células de mamíferos, e apenas uma fracção limitada de siRNAs desenhadas aleatoriamente é funcional. Além disso, os efeitos silenciosos fora do alvo surgem quando o siRNA tem uma complementaridade parcial na região de sementes com genes não intencionais. Aqui, com base na maquinaria RNAi, descrevemos o design racional de siRNAs funcionais, com efeito fora do alvo reduzido, que são esperados para derrubar um gene alvo-especificamente.
Declaração de conflito de interesses
os autores declaram que a investigação foi realizada na ausência de quaisquer relações comerciais ou financeiras que possam ser interpretadas como um potencial conflito de interesses.
Agradecimentos
Este trabalho foi parcialmente apoiado por subsídios do Ministério da Educação, Cultura, Desporto, Ciência e Tecnologia do Japão (MEXT), e a Célula de Inovação do Projeto (MEXT), e Núcleo de Projeto de Pesquisa para a Universidade Privada; correspondência fundo de subsídio para Kumiko Ui-Tei.
Amarzguioui, M., and Prydz, H. (2004). Um algoritmo para a selecção de sequências de siRNA funcionais. Bioquímica. Biophys. Res. Commun. 316, 1050–1058.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Birmingham, A., Almeida, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S., e Khvorova, A. (2006). 3 ‘ UTR seed matches, but not overall identity, are associated with RNAi off-targets. Conversao. Methods 3, 199-204.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Castanotto, D., e Rossi, J. J. (2009). As promessas e armadilhas do RNA-interference-based therapeutics. Nature 457, 426-433.
Pubmed Abstract / Pubmed texto completo | CrossRef texto completo
Chalk, A. M., and Sonnhammer, E. L. L. (2008). busca de especificidade siRNA incorporando dados de tolerância inadequados. Bioinformática 24, 1316-1317.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Cui, W., Ning, J., Naik, U. P., e Duncan, M. K. (2004). OptiRNAi, e RNAi design tool. Computa. Methods Programs Biomed. 75, 67–73.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Ding, Y., Chan, C. Y., and Lawrence, C. E. (2004). Sfold web server for statistical folding and rational design of nucleic acids. Nucleic Acids Res. 32, W135–W141.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K., e Tuschl, T. (2001). As Duplexes de 21 nucleótidos RNAs mediam a interferência de ARN em células de mamíferos em cultura. Nature 411, 494-498.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Elbashir, W. M., Harborth, J.; Weber, K., e Tuschl, T. (2002). Análise da função genética em células somáticas de mamíferos utilizando pequenas RNAs interferentes. Methods 26, 199-213.
Pubmed Abstract / Pubmed texto completo | CrossRef texto completo
Frank, F., Sonenberg, N. ,and Nagar, B. (2010). Base estrutural para o reconhecimento específico da base de 5′-nucleótidos do ARN-guia pelo AGO2 humano. Nature 465, 818-822.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S., e Li, Tongbin. (2008). siDRM: e ferramenta de design siRNA online eficaz e geralmente aplicável. Bioinformatics 24, 2405-2406.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lopes, L. P., e Bartel, D. P. (2007). MicroRNA direccionando especificidade em mamíferos: determinantes para além do emparelhamento de sementes. Mol. Cela 27, 91-105.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R., e Hannon, G. J. (2001). Argonaute 2 ligação entre análises genéticas e bioquímicas de RNAi. Science 293, 1146-1150.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Henschel, A., Buchholz, F., e Habermann, B. (2004). DEQOR: uma ferramenta baseada na web para o design e controle de qualidade de siRNAs. Nucleic Acids Res. 32, W113–W120.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Horn, T., Sandmann, T., e Boutros, M. (2010). Design and evaluation of genome-wide libraries for RNA interference screens. Genoma Biol. 11, R61.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Jackson, A. L., Bartz, S. R., Abrigo, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., e Linsley, P. S. (2003). O perfil de expressão revela regulação genética fora do alvo pela RNAi. Conversao. Biotechnol. 21, 635–637.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M.; Cleary, M., Lopes, L., e Linsley, P. S. (2006). Generalized siRNA “off-target” transcript silencing mediated by seed region sequence complementary. RNA 12, 1179-1187.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Ketting, R. F. (2011). Os muitos rostos de RNAi. Desenvolvimento. Cela 15, 148-161.
CrossRef Texto Completo
Khvorova, A., Reynolds, A., e Jayasena, S. D. (2003). As siRNAs funcionais e as miRNAs exibem viés de fio. Cela 115, 209-216.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Ladunga, I. (2007). Silenciamento genético mais completo por menos siRNAs: design otimizado transparente e assinatura Biofísica. Nucleic Acids Res. 35, 433-440.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Leuschner, P. J., Ameres, S. L., Kueng, S. e Martinez, J. (2006). Clivagem da cadeia de passageiros siRNA durante a montagem RISC em células humanas. EMBO Rep. 7, 314-320.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Lewis, B. P., Burge, C. B., e Bartel, D. P. (2005). Emparelhamento de sementes conservado, muitas vezes ladeado por adenosina, indica que milhares de genes humanos são alvos microRNA. Cela 120, 15-20.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Lopes, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M.; Castelo, J., Bartel, D. P., Linsley, P. S., Johnson, J. M. (2005). A análise de Microarray mostra que alguns microRNAs baixam a regulamentação de um grande número de mRNAs-alvo. Nature 433, 769-773.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Lu, Z. J., and Mathews, D. H. (2008). Uma selecção eficiente de siRNA usando hibridização termodinâmica. Nucleic Acid Res. 36, 640-647.
CrossRef Texto Completo
Ma, J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T., e Patel, D. J. (2005). Base estrutural para o reconhecimento específico de 5’de ARN-guia pela proteína A. fulgidus piwi. Nature 434, 666-670.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Martinez, J., Patkaniowska, A., Férias, H., Luhrmann, R., e Tuschl, T. (2002). SiRNAs antissenso de cadeia simples orientam a clivagem de ARN alvo em RNAi. Cela 110, 563-574.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., e Zamore, P. D. (2005). A clivagem Passenger-strand facilita a montagem de siRNA em complexos enzimáticos RNAi contendo Ago2. Cela 123, 607-620.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Naito, Y., Yoshimura, J., Morishita, S., e Ui-Tei, K. (2009). siDirect 2. 0: software atualizado para o projeto de siRNA funcional com efeito off-target dependente de sementes reduzido. BMC Bioinformatics 10, 392.
CrossRef texto completo
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M., and Kim, Y. J. (2008). AsiDesigner: exon-based siRNA design server considering alternative splicing. Nucleic Acid Res. 36, W97–W103.
CrossRef Texto Completo
Rand, T. A., Petersen, S., Du, F., e Wang, X. (2005). Argonaute2 clives the anti-guide strand of siRNA during RISC activation. Cela 123, 621-629.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Reynolds, A., Leake, D., Boese, P., Scaringe, S., Marshall, W. S., e Khvorova, A. (2004). Desenho siRNA racional para interferência de ARN. Conversao. Biotechnol. 22, 326–330.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N., e Zamore, P. D. (2003). Assimetria na montagem do complexo enzimático RNAi. Cela 115, 199-208.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Shah, J. K., Garner, H. R., Branco, M. A., Vergonhas, D. S., e Minna, J. D. (2007). sIR: siRNA information resource, a web-based tool for siRNA sequence design and analysis and an open access siRNA database. BMC Bioinformatics 8, 178. doi:10.1186/1471-2105-8-178
CrossRef Texto Completo
Interface do usuário-Tei, K., Naito, Y., Nishi, K., Juni, A., e Saigo, K. (2008). Estabilidade termodinâmica e emparelhamento de base Watoson-Crick no duplex seed são determinantes da eficiência do efeito off-target baseado em sRNA. Nucleic Acids Res. 36, 7100-7109.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Interface do usuário-Tei, K., Naito, Y. Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R., e Saigo, K. (2004). Guidelines for the selection of highly effective siRNA sequences for mammalian and chick RNA interference. Nucleic Acids Res. 32, 936-948.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Vert, J.-P., Foveau, N., Lajaunie, C., e Vandenbrouck, Y. (2006). Um modelo preciso e interpretável para a previsão de eficácia siRNA. BMC Bioinformatics 7, 520. doi:10.1186/1471-2105-7-520
CrossRef Texto Completo
Yuan, B., Latek, R., Rossbach, M., Tuschl, T., e Lewitter, F. (2004). siRAN selection server: an automatic siRNA oligonucleotide prediction server. Nucleic Acids Res. 32, W130–W134.
Pubmed Resumo | Pubmed Texto Completo | CrossRef Texto Completo
Yuan, Y. R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T., e Patel, D. J. (2005). Crystal structure of A. aeolicus argonaute, a site-specific DNA-guided endoribonuclease, provides insights into RISC-mediated mRNA cleavage. Mol. Cela 19, 405-419.
Pubmed Abstract / Pubmed Texto Completo / CrossRef Texto Completo