- Introducción
- Diseño optimizado de siRNA
- PASO 1: Selección de la Secuencia Funcional de ARNip
- PASO 2: Selección de Secuencias de ARNip con Efectos Fuera del Objetivo Reducidos
- PASO 3: Eliminación de Genes cercanos a la Diana Emparejados Casi Perfectos
- Software de diseño siRNA
- Conclusión
- Declaración de Conflicto de Intereses
- Reconocimientos
Introducción
La interferencia de ARN (ARNi) es una técnica ampliamente utilizada por la cual el ARN de interferencia pequeño (siRNA) regula a la baja un gen diana específico con una secuencia complementaria perfecta, y promete usarse en aplicaciones terapéuticas para enfermedades humanas (Castanotto y Rossi, 2009; Karting, 2011). Aunque el ser humano tiene más de 20.000 genes, es deseable utilizar el siRNA, que es altamente funcional y no tiene efectos en ningún otro gen que no sea su objetivo específico. En este artículo, revisamos un método optimizado para diseñar siRNA basado en el mecanismo del ARNi. Además, presentamos los sitios web abiertos al público para seleccionar secuencias de ARNip.
Diseño optimizado de siRNA
Los dúplex de ARN de 21 nucleótidos (nt) con voladizos de 2 nt 3′ (siRNA) se utilizan generalmente para experimentos con ARNi. Tras la entrega en las células, los ARN SIR se incorporan al complejo silenciador inducido por ARN (RISC) como ARN de doble cadena. RISC es el complejo efector que contiene proteína Argonauta (Ago) con actividad cortadora (Hammond et al., 2001; Martínez et al., 2002). La hebra guía siRNA que contiene el extremo 5’termodinámicamente menos estable es retenida preferentemente por RISC (Khvorova et al., 2003; Schwarz et al., 2003; Ui-Tei et al., 2004). Las hebras de pasajeros de la mayoría de los ARNIS de doble cadena cargados en RISC son escindidas por la proteína Ago2 y degradadas(Matranga et al., 2005; Rand et al., 2005; Leuschner et al., 2006). Los pares de hebras guía retenidas apuntan al ARNm con una secuencia perfectamente complementaria, y lo reprimen por escisión de la proteína Ago2 en la posición 10 del nucleótido de la hebra guía del ARNip(Elbashir et al., 2001; Hammond et al., 2001; Martínez et al., 2002). Sin embargo, una evidencia acumulada de experimentos de todo el genoma indica que un gran número de ARNM con complementariedades parciales a la hebra guía también se reducen (Jackson et al., 2003, 2006; Lim et al., 2005; Birmingham et al., 2006; Ui-Tei et al., 2008). Este fenómeno se conoce como efecto fuera del objetivo dependiente de la semilla y se observa preferiblemente en ARNm 3 ‘ UTRs. Se sabe que el mecanismo de reconocimiento del objetivo de este efecto fuera del objetivo es similar al del silenciamiento génico mediado por miARN (Lewis et al., 2005; Lim et al., 2005; Grimson et al., 2007). Las transcripciones con secuencias complementarias a la región semilla posicionada 2-8 desde el terminal de 5’ se reducen principalmente. Se sabe que la región semilla está situada en la superficie de Ago en una forma cuasi-helicoidal para servir como sitio de entrada o nucleación para pequeños ARN en los RISC (Ma et al., 2005; Yuan et al., 2005). Por lo tanto, la región de la semilla primero identifica los ARNm objetivo, y posteriormente forma un emparejamiento perfecto de la base con el ARNm objetivo previsto e induce ARNi mediante Ago2.
Basado en el mecanismo del ARNi, un ARNip específico del gen diana se considera seleccionable de acuerdo con los tres pasos siguientes.
PASO 1: Selección de la Secuencia Funcional de ARNip
Las eficiencias de derribo de los ARNip se revelan altamente dependientes de sus secuencias. Defendimos la regla empírica que prescribía las características de los ARNIS altamente funcionales (Ui-Tei et al., 2004), dicha regla se denomina regla Ui-Tei (Figura 1). El siRNA seleccionado por la regla Ui-Tei cumple simultáneamente las siguientes cuatro condiciones: (1) A o U en la posición 1 desde el extremo 5′ de la hebra guía del ARNip, (2) G o C en la posición 19, (3) riqueza de AU (AU ≥4) en las posiciones 1-7, y (4) sin estiramiento largo de GC ≥10. A excepción de (4), nuestra regla indica que el siRNA funcional tiene estabilidad asimétrica en terminales de 5′ y 3′. Nuestra validación experimental mediante el ensayo del reportero de luciferasa mostró que el 98% de los ARNip que satisfacían las condiciones anteriores redujeron la expresión del reportero de luciferasa por debajo del 33% (Ui-Tei et al., 2004). Otros grupos también demostraron las reglas de siRNAs altamente funcionales conocidas como regla de Reynolds (Reynolds et al., 2004) y la regla de Amarzguioui (Amarzguioui y Prydz, 2004), como se resume en la Figura 1. Estas reglas también mostraron claramente que los siRNAs funcionales son asimétricos: una hebra de ARN con terminal inestable de 5′ era efectiva como hebra guía. Además, común en estas reglas, el término 5′ de la hebra guía funcional del ARNip era preferible a ser A o U. Se reveló más tarde que los resultados reflejaban las características estructurales de la Ago2 humana (Frank et al., 2010). La estructura cristalina de un dominio MEDIO (medio) de los experimentos de titulación AGO2 y RMN en humanos mostró que los monofosfatos de nucleótidos, AMP y UMP, se unen con una afinidad hasta 30 veces mayor que CMP o GMP, proporcionando evidencia estructural de interacciones específicas de nucleótidos en el dominio MEDIO de las proteínas eucarióticas AGO.FIGURA
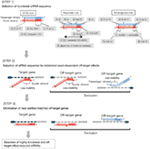
Gráfico 1 Representación esquemática para seleccionar ARNIS funcionales y con efecto reducido fuera del objetivo. Selección de siRNA altamente funcional por reglas Ui-Tei, Reynolds, Amarzguioui, o la combinación de ellas (PASO 1). Selección de ARNIS con baja estabilidad en los dúplex objetivo de siembra (PASO 2). Eliminación de ARNIS con secuencias casi perfectas que coinciden con genes no diana (PASO 3). En cada regla, la posición del nucleótido indica el número de nucleótidos contados a partir del terminal 5′ de la hebra guía.  :
:  A / U en la posición 1.
A / U en la posición 1.  G / C en la posición 19.
G / C en la posición 19.  4 a 7 A / Us en las posiciones 1-7.
4 a 7 A / Us en las posiciones 1-7. 
 :
:  Contenido de CG (30% -52%).
Contenido de CG (30% -52%).  A / U ≥ 3 en las posiciones 1-5.
A / U ≥ 3 en las posiciones 1-5.  Ausencia de repeticiones internas.
Ausencia de repeticiones internas.  A en la posición 1.
A en la posición 1.  A en la posición 17.
A en la posición 17.  U en la posición 10.
U en la posición 10.  No G / C en la posición 1.
No G / C en la posición 1.  No G en la posición 7.
No G en la posición 7. 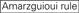 :
:  Asimetría en la estabilidad de los extremos dúplex (medida como el diferencial A / U de los tres pares base terminales en cada extremo del dúplex).
Asimetría en la estabilidad de los extremos dúplex (medida como el diferencial A / U de los tres pares base terminales en cada extremo del dúplex).  G o C en la posición 19.
G o C en la posición 19.  A o U en la posición 1.
A o U en la posición 1.  A o U en la posición 14.
A o U en la posición 14.  No U en la posición 19.
No U en la posición 19.  No G en la posición 1.
No G en la posición 1.
PASO 2: Selección de Secuencias de ARNip con Efectos Fuera del Objetivo Reducidos
Para evitar efectos fuera del objetivo dependientes de la semilla, un enfoque puede ser seleccionar la cadena guía de ARNip cuya secuencia de semilla no es complementaria a ninguna secuencia en el 3′ UTR de todos los genes no objetivo. Sin embargo, se ha demostrado que este enfoque es imposible, porque los ARNR humanos con la secuencia de semillas de siete nt más infrecuente aún tenían complementariedades de semillas con varios ARNm no dirigidos. Por lo tanto, hemos buscado las reglas que rigen la capacidad de los ARNIS para inducir el efecto fuera del objetivo dependiente de la semilla, y revelamos que la eficiencia del efecto fuera del objetivo está altamente correlacionada con la estabilidad termodinámica del dúplex formado entre la región de la semilla de la hebra guía de arNIS y su ARNm objetivo (Ui-Tei et al., 2008). La temperatura de fusión (Tm), uno de los parámetros termodinámicos para la formación de ARN dúplex, mostró una fuerte correlación positiva con la inducción de efectos fuera del objetivo dependientes de la semilla. Por lo tanto, la selección de siRNAs con Tm baja del dúplex semilla-objetivo debe minimizar el silenciamiento fuera del objetivo dependiente de la semilla (Figura 1). La Tm de 21,5°C puede servir como punto de referencia, que discrimina las secuencias de semillas casi fuera de objetivo de las secuencias positivas fuera de objetivo. Además, dado que el efecto fuera del objetivo puede ser causado no solo por la hebra guía, sino también por la hebra pasajera, los ARNR cuya Tm de objetivo de semilla es suficientemente baja para ambas hebras son favorables.
PASO 3: Eliminación de Genes cercanos a la Diana Emparejados Casi Perfectos
Incluso cuando el valor de Tm del dúplex semilla-objetivo es suficientemente bajo, el silenciamiento del gen objetivo aún puede tener lugar si la región no semilla es completamente complementaria. Por lo tanto, en el tercer paso, se eliminaron los ARNR que tenían coincidencias casi perfectas con cualquier otra transcripción no dirigida (Figura 1).
Software de diseño siRNA
Presentamos el software de diseño siRNA, siDirect 2.0 (http://siDirect2.RNAi.jp/; Figura 2), que proporciona un software de diseño de ARNip funcional y específico para el objetivo de acuerdo con los procedimientos mencionados anteriormente (Naito et al., 2009). En el parámetro predeterminado, se puede seleccionar la regla Ui-Tei de siRNAs que satisfaga. Cuando los ARNr funcionales candidatos podrían formar duplicados de objetivo semilla con valores de Tm por debajo de 21,5°C, y sus regiones de 19 nt que abarcan las posiciones 2-20 de ambos filamentos tienen al menos dos desajustes con cualquier otra transcripción no objetivo, siDirect 2.0 puede diseñar al menos un ARNr calificado para >94% de secuencias de ARNm humanos en RefSeq.FIGURA

Gráfico 2 Vistas de pantalla del software de diseño siRNA siDirect 2.0. A) Página superior (http://siDirect2.RNAi.jp/). B) Parámetros opcionales para el diseño de siRNA. C) Página de resultados. D) Lista detallada de candidatos no seleccionados con coincidencias casi perfectas. La alineación entre cada transcripción fuera de objetivo y la secuencia de siRNA visualiza las posiciones de los desajustes.
Otros programas informáticos para seleccionar SIRNAS funcionales estaban abiertos al público, como se muestra en la Tabla 1. En muchos de ellos, la regla Ui-Tei (Ui-Tei et al., 2004), regla de Reynolds (Reynolds et al., 2004), la regla de Amarzguioui (Amarzguioui y Prydz, 2004), la regla de Tuschl (Elbashir et al., 2002), y la combinación de ellos se utilizó con frecuencia y ampliamente. Para eliminar los genes no objetivo casi perfectos, se utilizó la búsqueda de EXPLOSIONES para la búsqueda de homología en varios programas informáticos. Sin embargo, dado que la búsqueda de EXPLOSIONES no es tan precisa para secuencias cortas como siRNAs, siDirect, WU-BLAST y Bowtie, que son motores de búsqueda de homología altamente precisos para secuencias cortas se utilizan a menudo. Entre ellos, siDirect 2.0 puede traer los resultados más precisos. Además, algunos de los programas tienen en cuenta las características adicionales, como la estructura secundaria de ARNm (Ladunga, 2007; Lu y Mathews, 2008), el empalme alternativo (Park et al., 2008), o la secuencia de motivos por la que podría ocurrir la respuesta inmune de un virus de ARN (Gong et al., 2008). Estas características no se consideran en siDirect 2.0. Por lo tanto, para tener en cuenta estas características, los SIRNAS comúnmente seleccionados por siDirect 2.0 y otros programas de software apropiados pueden producir un resultado óptimo.
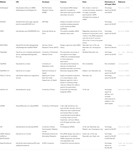
Cuadro 1 Pequeños programas de software de diseño de ARN de interferencia.
Además, es prácticamente importante considerar el uso de dos o más ARNip dirigidos a diferentes sitios en un gen objetivo previsto, ya que se supone que los efectos de derribo de un gen objetivo previsto son comunes, pero es probable que los efectos fuera del objetivo sean diferentes entre los ARNip.
Conclusión
Se sabe que la eficacia de cada siRNA varía ampliamente dependiendo de su secuencia en células de mamíferos, y solo una fracción limitada de siRNAs diseñados aleatoriamente es funcional. Además, los efectos silenciadores fuera del objetivo surgen cuando el siRNA tiene complementariedad parcial en la región de la semilla con genes no deseados. Aquí, basado en la maquinaria de ARNi, describimos el diseño racional de ARNIS funcionales, con efecto fuera del objetivo reducido, que se espera que derriben un gen objetivo, específicamente.
Declaración de Conflicto de Intereses
Los autores declaran que la investigación se realizó en ausencia de relaciones comerciales o financieras que pudieran interpretarse como un conflicto de intereses potencial.
Reconocimientos
Este trabajo fue apoyado parcialmente por subvenciones del Ministerio de Educación, Cultura, Deportes, Ciencia y Tecnología de Japón (MEXT), y el Proyecto de Innovación Celular (MEXT), y el Proyecto de Investigación Central para Universidad Privada; subsidio de fondo de contrapartida a Kumiko Ui-Tei.
Amarzguioui, M., and Prydz, H. (2004). Un algoritmo para la selección de secuencias de ARNip funcionales. Bioquímica. Biophys. Res. Commun. 316, 1050–1058.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Birmingham, A., Anderson, E. M., Reynolds, A., Ilsley-Tyree, D., Leake, D., Fedorov, Y., Baskerville, S., Maksimova, E., Robinson, K., Karpilow, J., Marshall, W. S., and Khvorova, A. (2006). Las coincidencias de semilla UTR de 3′, pero no la identidad general, están asociadas con ARNi fuera de los objetivos. NAT. Methods 3, 199-204.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Castanotto, D., and Rossi, J. J. (2009). Las promesas y trampas de la terapia basada en ARN-interferencia. Nature 457, 426-433.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Chalk, A. M., and Sonnhammer, E. L. L. (2008). Búsqueda de especificidad de siRNA que incorpora datos de tolerancia de desajuste. Bioinformática 24, 1316-1317.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Cui, W., Ning, J., Naik, U. P., and Duncan, M. K. (2004). Herramienta de diseño OptiRNAi y RNAi. Comput. Métodos Programas Biomed. 75, 67–73.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Ding, Y., Chan, C. Y., and Lawrence, C. E. (2004). Servidor web Sfold para plegado estadístico y diseño racional de ácidos nucleicos. Ácidos nucleicos Res. 32, W135-W141.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Elbashir, S. M., Harborth, J., Lendeckel, W., Yalcin, A., Weber, K., and Tuschl, T. (2001). Los ARN dúplex de 21 nucleótidos median la interferencia de ARN en células de mamíferos cultivadas. Nature 411, 494-498.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Elbashir, W. M., Harborth, J., Weber, K., and Tuschl, T. (2002). Análisis de la función génica en células somáticas de mamíferos utilizando ARN pequeños que interfieren. Methods 26, 199-213.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Frank, F., Sonenberg, N., y Nagar, B. (2010). Base estructural para el reconocimiento específico de la base de nucleótidos 5’del ARN guía por AGO2 humano. Nature 465, 818-822.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Gong, W., Ren, Y., Zhou, H., Wang, Y., Kang, S., and Li, Tongbin. (2008). siDRM: herramienta de diseño de siRNA en línea eficaz y de aplicación general. Bioinformática 24, 2405-2406.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Grimson, A., Farh, K. K., Johnston, W. K., Garrett-Engele, P., Lim, L. P., and Bartel, D. P. (2007). microRNA targeting specificity in mammals: determinants beyond seed pairing. Mol. Celda 27, 91-105.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Hammond, S. M., Boettcher, S., Caudy, A. A., Kobayashi, R., and Hannon, G. J. (2001). Argonaute 2 enlace entre análisis genéticos y bioquímicos de ARNi. Science 293, 1146-1150.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Henschel, A., Buchholz, F., y Habermann, B. (2004). DEQOR: una herramienta basada en la web para el diseño y control de calidad de siRNAs. Ácidos nucleicos Res. 32, W113-W120.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Horn, T., Sandmann, T., y Boutros, M. (2010). Diseño y evaluación de bibliotecas genómicas para pantallas de interferencia de ARN. Genome Biol. 11, R61.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Jackson, A. L., Bartz, S. R., Shelter, J., Kobayashi, S. V., Burchard, J., Mao, M., Li, B., Cavet, G., and Linsley, P. S. (2003). El perfil de expresión revela la regulación de genes fuera del objetivo por ARNi. NAT. Biotechnol. 21, 635–637.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Jackson, A. L., Burchard, J., Schelter, J., Chau, B. M., Cleary, M., Lim, L., and Linsley, P. S. (2006). Silenciamiento generalizado de transcripción de siRNA «fuera de objetivo» mediado por la complementariedad de la secuencia de la región semilla. ARN 12, 1179-1187.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Karting, R. F. (2011). Las muchas caras del ARNi. Dev. Celda 15, 148-161.
Texto completo Cruzado
Khvorova, A., Reynolds, A., and Jayasena, S. D. (2003). Los siRNAs y miRNAs funcionales exhiben sesgo de cadena. Celda 115, 209-216.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Ladunga, I. (2007). Silenciamiento génico más completo con menos ARNIS: diseño optimizado transparente y firma biofísica. Nucleic Acids Res. 35, 433-440.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Leuschner, P. J., Ameres, S.L., Kueng, S., and Martinez, J. (2006). Escisión de la cadena de pasajeros de siRNA durante el ensamblaje de RISC en células humanas. EMBO Rep. 7, 314-320.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Lewis, B. P., Burge, C. B., and Bartel, D. P. (2005). El emparejamiento de semillas conservadas, a menudo flanqueado por adenosina, indica que miles de genes humanos son blancos de microRNA. Celda 120, 15-20.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Lim, L. P., Lau, N. C., Garrett-Engele, P., Grimson, A., Schelter, J. M., Castle, J., Bartel, D. P., Linsley, P. S., and Johnson, J. M. (2005). El análisis de microarray muestra que algunos microRNAs regulan a la baja un gran número de ARNM objetivo. Nature 433, 769-773.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Lu, Z. J., and Mathews, D. H. (2008). Selección eficiente de siRNA mediante termodinámica de hibridación. Nucleic Acid Res. 36, 640-647.
Texto completo Cruzado
Ma,J.-B., Yuan, Y. R., Meister, G., Pei, Y., Tuschl, T., and Patel, D. J. (2005). Base estructural para el reconocimiento específico del extremo 5’del ARN guía por la proteína A. fulgidus piwi. Nature 434, 666-670.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Martinez, J., Patkaniowska, A., Urlaub, H., Luhrmann, R., and Tuschl, T. (2002). Los SIRNAS antisentido de una sola cadena guían la escisión del ARN objetivo en el ARNI. Celda 110, 563-574.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Matranga, C., Tomari, Y., Shin, C., Bartel, D. P., and Zamore, P. D. (2005). La hendidura de la cadena de pasajeros facilita el ensamblaje de ARNi en complejos enzimáticos ARNi que contienen Ago2. Celda 123, 607-620.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Naito, Y., Yoshimura, J., Morishita, S., and Ui-Tei, K. (2009). siDirect 2.0: software actualizado para el diseño de siRNA funcional con efecto descentrado dependiente de semillas reducido. Bioinformática BMC 10, 392.
Texto completo Cruzado
Park, Y.-K., Park, S.-M., Choi, Y.-C., Lee, D., Won, M., and Kim, Y. J. (2008). AsiDesigner: servidor de diseño siRNA basado en exones que considera empalmes alternativos. Ácido nucleico Res. 36, W97-W103.
Texto completo Cruzado
Rand, T. A., Petersen, S., Du, F., and Wang, X. (2005). Argonaute2 escinde la hebra antiguía del ARNip durante la activación RISC. Celda 123, 621-629.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Reynolds, A., Leake, D., Boese, Q., Scaringe, S., Marshall, W. S., and Khvorova, A. (2004). Diseño racional de siRNA para interferencia de ARN. NAT. Biotechnol. 22, 326–330.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Schwarz, D. S., Hutvangner, G., Du, T., Xu, Z., Aronin, N., and Zamore, P. D. (2003). Asimetría en el ensamblaje del complejo enzimático ARNi. Celda 115, 199-208.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Shah, J. K., Garner, H. R., White, M. A., Shames, D. S., and Minna, J. D. (2007). sIR: siRNA information resource, una herramienta basada en la web para el diseño y análisis de secuencias de siRNA y una base de datos de siRNA de acceso abierto. Bioinformática BMC 8, 178. doi:10.1186/1471-2105-8-178
Texto completo de CrossRef
Ui-Tei, K., Naito, Y., Nishi, K., Juni, A., and Saigo, K. (2008). La estabilidad termodinámica y el emparejamiento de bases Watoson-Crick en el dúplex de semillas son los principales determinantes de la eficiencia del efecto fuera de objetivo basado en sRNA. Nucleic Acids Res. 36, 7100-7109.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Ui-Tei, K., Naito, Y., Takahashi, F., Haraguchi, T., Ohki-Hamazaki, H., Juni, A., Ueda, R., and Saigo, K. (2004). Directrices para la selección de secuencias de ARN SIR altamente eficaces para la interferencia de ARN de mamíferos y pollitos. Nucleic Acids Res. 32, 936-948.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Vert, J.-P., Foveau, N., Lajaunie, C., and Vandenbrouck, Y. (2006). Un modelo preciso e interpretable para la predicción de la eficacia del siRNA. Bioinformática BMC 7, 520. doi:10.1186/1471-2105-7-520
Texto completo de CrossRef
Yuan, B., Latek, R., Rossbach, M., Tuschl, T., and Lewitter, F. (2004). Servidor de selección de siRAN: un servidor automatizado de predicción de oligonucleótidos siRNA. Ácidos nucleicos Res. 32, W130-W134.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado Completo
Yuan, Y.-R., Pei, Y., Ma, J. B., Kuryavyi, V., Zhandina, M., Meister, G., Chen, H. Y., Dauter, Z., Tuschl, T., and Patel, D. J. (2005). La estructura cristalina de A. aeolicus argonaute, una endoribonucleasa guiada por ADN específica del sitio, proporciona información sobre la escisión del ARNm mediada por RISC. Mol. Celda 19, 405-419.
Resumen de Pubmed / Texto Completo de Pubmed / Texto Cruzado